Frontiers in Genetics
Introdução
Muitas características são reguladas e coordenadas por múltiplos genes e condições ambientais. Na natureza, tais traços evoluem continuamente sob pressões de seleção local e processos neutros, levando a uma rica diversidade de variedades fenotípicas e estratégias ambientais de sobrevivência. Já Dobzhansky (1964) descreveu a rica diversidade do mundo vivo como o resultado da diversidade genética, da heterogeneidade ambiental e das adaptações que evoluem na interação entre a biologia molecular e a biologia do organismo. Os mecanismos moleculares em evolução têm sido particularmente difíceis de caracterizar. Em primeiro lugar, precisamos de ligar o genótipo ao fenótipo. Esta ligação consiste em extensas e intrincadas redes de interacção genética (Ayroles et al., 2009; Lehner, 2013). Em segundo lugar, precisamos identificar as mudanças moleculares responsáveis pelas adaptações fenotípicas. Isto depende da elucidação da variação genética e das alterações genéticas que podem ocorrer em qualquer parte das redes de interação genética (por exemplo, Edwards et al., 2009).
A manifestação da variação genética nas redes de genes é muito complexa. Alterações em genes únicos podem afectar a actividade e mesmo a topologia de toda a rede genética (p. ex., Knight et al., 2006). Genes e redes de interação genética são frequentemente pleiotrópicos e regulam vários traços e processos, implicando que mudanças em um único gene podem ser manifestadas em vários traços (Stearns, 2010). Por outro lado, variações alélicas em muitos genes podem contribuir para a variação de um determinado traço fenotípico (Manolio et al., 2009). Além disso, a epistasia é generalizada, implicando que as variações alélicas em múltiplos loci podem afetar os efeitos fenotípicos uns dos outros (Phillips, 2008; Mackay e Moore, 2014; Moore e Williams, 2015). Estas considerações sobre a base genética da evolução não são novas e têm sido estudadas há várias décadas (por exemplo, Wagner e Altenberg, 1996). A genética directa e a genética inversa têm tido grande sucesso na elucidação das funções de genes únicos ou mutações para uma característica particular (Nagy et al., 2003). Estas técnicas, no entanto, são limitantes ao estudar a complexidade das redes de interação molecular subjacentes a um fenótipo, ou os mecanismos moleculares da evolução de traços complexos.
Os desenvolvimentos na tecnologia da genômica têm sido um grande impulso para a nossa capacidade de estudar as complexidades genéticas dos traços fenotípicos e sua evolução (Stapley et al., 2010). A combinação destas técnicas com as abordagens da genética clássica permite-nos avaliar a funcionalidade da variação genética dos traços fenotípicos (Storz and Wheat, 2010). Os primeiros estudos genómicos sobre vários organismos modelo enfatizaram que as adaptações evolutivas, mesmo para condições ambientais específicas, geralmente governam muitos genes ou loci, assim como a regulação dinâmica dos padrões de expressão genética (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). Na década seguinte, muitos estudos utilizaram a genômica para identificar genes e proteínas que estavam contribuindo para traços particulares e interações ecológicas. Inicialmente, os custos e o tempo necessários para sequenciar um único genoma ainda eram altamente restritivos. O sequenciamento da próxima geração, no entanto, tornou possível sequenciar os genomas de muito mais espécies, e muito mais indivíduos por espécie. Este é um recurso formidável para estudar a evolução, pois nos permite, pela primeira vez, mapear as mudanças em todo o genoma durante a evolução.
A tecnologia genómica tem um enorme potencial para melhorar os nossos conhecimentos sobre os processos evolutivos. Abordagens comparativas têm sido aplicadas para mapear as mudanças nas sequências genómicas ou redes de interacção genética em longos períodos de tempo evolutivos (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Selecção experimental ou abordagens de evolução experimental, seguidas de transcriptómica ou sequenciação genómica, têm sido usadas para mapear alterações evolutivas em escalas de tempo muito mais curtas (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Estes estudos mostraram, por exemplo, como duplicações de genes, mutações e fortes divergências de seqüência em pequenos subconjuntos de genes podem ter um profundo impacto sobre a atividade transcripcional de grandes redes de interação genética e múltiplos traços fenotípicos.
Nesta perspectiva, apresento os recentes achados e desenvolvimentos sobre a base genômica da evolução, usando a imunidade evolutiva como um estudo de caso. Imunidade é um traço que evolui rapidamente, tornando possível o estudo da base genómica dos processos evolutivos (Obbard et al., 2009; Sironi et al., 2015). Além disso, as redes moleculares em imunidade têm sido bastante bem caracterizadas devido à sua importância para a saúde humana (Schadt, 2009; Lazzaro e Schneider, 2014; Zak et al., 2014). Em primeiro lugar, resumi muito brevemente o nosso entendimento actual sobre as redes moleculares de respostas imunitárias inatas e os processos de selecção que actuam nas respostas imunitárias. Depois, descrevo as mudanças genómicas, associadas com o ganho, a perda e a modulação de aspectos particulares das respostas imunitárias em Drosophila. Finalmente, proponho direções futuras para estudar a arquitetura genética de traços complexos e processos evolutivos.
Respostas imunológicas
O sistema imunológico consiste de uma combinação de processos fisiológicos que atuam em conjunto na defesa contra patógenos e parasitas. A imunidade inata é uma característica antiga que pode ser encontrada em todos os organismos multicelulares, enquanto que os vertebrados também possuem imunidade adquirida. Ambos os sistemas imunitários combinam componentes celulares e humorais: o componente celular compreende células especializadas que proporcionam uma função protectora. Isto inclui, classes de células sanguíneas para fagocitose de micróbios, encapsulação de corpos estranhos maiores ou reconhecimento de antígenos (na imunidade adquirida), e o revestimento do intestino com células epiteliais que formam uma barreira física e podem secretar compostos defensivos. O componente humoral consiste na liberação de fatores extracelulares que combatem os patógenos invasores, muitas vezes a partir de tecidos especializados ou populações celulares. Isto inclui a liberação de peptídeos antimicrobianos (AMPs) pelo fígado (ou o corpo adiposo em invertebrados) e células epiteliais, espécies reativas de oxigênio em células fagocitárias e epiteliais, e anticorpos de glóbulos brancos (em imunidade adquirida; Lemaitre e Hoffmann, 2007; Buchmann, 2014).
Redes complexas de interações moleculares coordenam as respostas imunológicas (Figura 1A). As mesmas vias são centrais para as respostas imunológicas dos invertebrados aos vertebrados, implicando em forte conservação dos elementos centrais das redes moleculares na imunidade (Silverman e Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). Uma variedade de moléculas receptoras pode reconhecer patógenos ou parasitas, por exemplo, com base em padrões moleculares associados a patógenos (por exemplo, lipopolissacáridos específicos de membranas bacterianas). Uma vez que os receptores são ativados, eles induzem vias específicas de transdução de sinal, tais como as vias Toll, Imd e Jak/Stat (revisado em Lemaitre e Hoffmann, 2007; Buchmann, 2014). Estas vias consistem em proteases, kinases, citocinas e outras proteínas que eventualmente ativam fatores de transcrição e co-fatores. A indução desses fatores de transcrição resulta na produção de moléculas de efeito humoral (por exemplo, AMPs) e pode induzir a proliferação e diferenciação das células envolvidas na imunidade. A produção de diferentes classes de células sanguíneas é um aspecto proeminente do componente celular da resposta imunológica, tanto na imunidade inata como na adquirida. Nestas células sanguíneas, as cascatas de transdução de sinal também são reguladas para induzir propriedades celulares e proteínas que efetivam a desobstrução do parasita. Para regular a força, especificidade, tempo e duração das respostas imunológicas, as redes moleculares são moduladas por citocinas, proteases e conversas cruzadas com outras vias de sinalização (Liew et al., 2005; Aggarwal e Silverman, 2008). Isto também inclui diversas redes reguladoras pós-transcripcionais (Ivanov e Anderson, 2013; Carpenter et al., 2014).
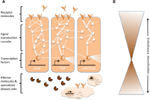
Figura 1. Representação esquemática das redes genéticas na imunidade. (A) Várias redes interligadas coordenam as respostas a um desafio imunológico. Estas redes consistem em proteínas (representadas por círculos) que interagem com cada uma delas numa cascata de transdução de sinal para regular a expressão dos factores de transcrição (representados por hexágonos). A ativação das vias de transdução do sinal central (por exemplo, IMD, Toll, ou Jak/Stat, indicado por linhas grossas entre proteínas) resulta na produção de moléculas efetoras, como peptídeos antimicrobianos (representados por símbolos em forma de torta) e na proliferação e diferenciação de células especializadas (sangue) (figuras em forma de nuvens). Moléculas extracelulares e membranas receptoras (figuras em forma de lua) induzem as vias de passagem. A actividade pode ser ainda modulada por muitas outras proteínas que interagem com as vias e conversam com outras vias e redes genéticas (indicadas pelas linhas finas entre as proteínas). (B) Os componentes centrais das redes genéticas na imunidade, por exemplo, os fatores de transcrição e as proteínas em contato direto com esses fatores de transcrição, são muitas vezes fortemente conservados através da phyla. A diversificação evolutiva é encontrada mais extensivamente em direção às periferias das redes.
Os motores das mudanças evolutivas nas respostas imunológicas são os efeitos combinados dos altos custos da aptidão física dos hospedeiros, os custos da imunidade, a rica diversidade de patógenos e parasitas que ameaçam os hospedeiros, e as dinâmicas corridas de armas co-evolucionárias entre hospedeiros e patógenos (Schmid-Hempel, 2003). Qualquer um ou todos estes aspectos podem operar no ambiente local do hospedeiro, e levar a fortes pressões de seleção. O tipo de seleção, no entanto, varia de direcional a purificação e equilíbrio, dependendo dos custos e benefícios que a população hospedeira experimenta em seu ambiente local. Por exemplo, um patógeno altamente virulento que invade uma comunidade local pode causar uma varredura seletiva ou seleção direcional para alelos de resistência particulares, já que apenas os hospedeiros com esses alelos podem contribuir para as próximas gerações. Alternativamente, uma comunidade diversificada ou co-evolutiva de patógenos pode conduzir a uma seleção dependente da freqüência ou do equilíbrio, favorecendo a manutenção da variação genética. A evolução das respostas imunológicas reflete tanto essas dinâmicas co-evolutivas com o parasita, quanto os custos fisiológicos e ecológicos do sistema imunológico (Kraaijeveld et al, 2002; Rolff e Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro e Little, 2009).
Base genómica da mudança evolutiva da imunidade Drosophila
Embora a maquinaria central das respostas imunitárias esteja fortemente conservada, vários componentes das redes moleculares alargadas podem evoluir rapidamente ou diversificar-se (Figura 1B). Em Drosophila, a rápida mudança evolutiva tem sido relatada para os receptores e os efetores da resposta imune (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Estas moléculas operam na interface entre o hospedeiro e o patógeno e, portanto, são cruciais para o reconhecimento pelo hospedeiro de um organismo invasor e para mediar os efeitos alvo e antagónicos da resposta imunológica sobre o patógeno. Ao mesmo tempo, o parasita está sob seleção para passar despercebido, para evitar ou mitigar os efeitos antagônicos da resposta imunológica. Portanto, a dinâmica da Red Queen é esperada para estas moléculas na interface, e aquelas que modulam as respostas imunológicas. Cada parte está tentando ganhar vantagem na corrida armamentista antagônica, provocando alterações recíprocas nas redes genéticas das partes. A diversificação nas moléculas receptoras, moduladoras e efetoras é realizada principalmente por duplicações genéticas e mudanças rápidas de seqüência (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Estamos estudando a resposta imunológica de Drosophila contra vespas parasitoides como um sistema modelo para entender a base genômica dos processos evolutivos. As larvas de Drosophila são hospedeiras de uma variedade de espécies de parasitoides que põem um ovo nestas larvas (Fleury et al., 2009). Assim que o ovo parasitoide eclode (∼2-4 dias após o ataque parasitoide, dependendo da espécie parasitoide e da temperatura), as larvas parasitoides começam a alimentar-se do hospedeiro e matam-no. Algumas espécies de Drosophila têm um mecanismo de defesa contra os parasitoides através de uma resposta imune inata, chamada encapsulação melanótica. Esta resposta imunitária consiste em componentes celulares e humorais que actuam em conjunto para sequestrar e matar o ovo do parasitoide. O ataque parasitoide desencadeia vias de transdução de sinal imune que induzem (i) a proliferação e diferenciação de duas classes de hemócitos (ou seja, células sanguíneas de insetos) que aderem ao ovo parasitoide e um ao outro, e (ii) a deposição de melanina no ovo parasitoide e na cápsula celular ao redor do ovo parasitoide (Lemaitre e Hoffmann, 2007). O hospedeiro tem que completar o encapsulamento completo e a melanização antes da eclosão do ovo parasitoide para sobreviver à infestação parasitoide.
Prior da era genómica, foram identificados vários genes que estavam envolvidos na resposta imunológica contra as vespas parasitoides. As vias Toll e Jak/Stat tinham sido identificadas como componentes centrais da proliferação e diferenciação de hemócitos, e a via da profenoloxidase para melanização (revista em Brennan e Anderson, 2004). Dois estudos de microarranjo foram então conduzidos para identificar genes adicionais potencialmente envolvidos no encapsulamento melanótico após o ataque de parasitoides. Esta abordagem destacou muitos genes adicionais que não haviam sido previamente associados à resposta imunológica contra parasitoides, e revelou seu tempo de ação (Wertheim et al., 2005; Schlenke et al., 2007). Os estudos revelaram vários grupos coordenados e funcionalmente coerentes de genes que foram temporariamente regulados para cima ou para baixo durante parte da resposta imunológica (Wertheim et al., 2005). Curiosamente, foi mostrado que os mecanismos de virulência de duas espécies de parasitoides diferiram em como interferiram na rede genética das respostas dos hospedeiros: uma espécie eliminou a ativação inicial de toda a rede, enquanto outra espécie visou o passo final na cascata (Schlenke et al., 2007).
Espécies de Drosophila diferem em grande parte na imunidade contra parasitoides. Algumas espécies são completamente susceptíveis aos parasitóides e isto foi relatado como uma deficiência imunológica (Eslin e Doury, 2006). Uma inspeção mais próxima, no entanto, revelou que a resistência aos parasitóides não é comumente compartilhada entre todas as espécies de Drosophila, mas é restrita a alguns clades. Mostramos que em um desses clades, o subgrupo melanogaster, o ganho evolutivo da resistência parasitóide estava associado ao ganho de um novo tipo de célula sangüínea, os lamelócitos, que também está restrito ao mesmo clade (Salazar-Jaramillo et al., 2014). Algumas espécies de Drosophila fora do subgrupo melanogaster também podem encapsular ovos parasitóides, mas parecem ter desenvolvido diferentes tipos de células sanguíneas para a resposta de encapsulamento (Havard et al., 2012; Márkus et al., 2015). A resposta imunológica contra parasitoides evoluiu independentemente em vários taxa de insetos, muitas vezes com mecanismos e tipos ligeiramente diferentes de células sangüíneas (Lavine and Strand, 2002). Assim, a evolução do sistema imunológico inato inclui a adição de novos componentes ou “módulos”. Isto levanta a questão de como o genoma muda durante a aquisição de um novo módulo.
Genômica comparativa revelou que, apesar do ganho de um novo tipo de célula sangüínea no subgrupo melanogaster, os genes que são conhecidos por diferenciação de lamelócitos são em grande parte conservados em toda a filogenia. Também as espécies que não produzem lamelócitos em resposta ao ataque de parasitoides possuem estes genes que são necessários para a diferenciação dos lamelócitos. Além disso, estes genes mostram pouca divergência ou assinaturas de seleção, enquanto que isso seria esperado para genes que obtiveram uma nova função. Isto indica que as vias de transdução de sinal existentes para diferenciação de hemócitos estão sendo moduladas pela rede de interação dos genes vizinhos para produzir um novo tipo de célula sangüínea no subgrupo melanogaster. Esta cooptação da via de proliferação de hemócitos do núcleo existente é provavelmente obtida pela adição de outros ou novos componentes à rede de interação do gene (Salazar-Jaramillo et al., 2014). Identificamos vários genes novos que surgiram na época da aquisição de lamelócitos e que são expressos diferentemente durante a resposta imunológica contra parasitoides, incluindo moléculas receptoras e proteases tipo serina (Salazar-Jaramillo et al., 2014). Hipotecamos que especialmente as endopeptidases do tipo serina podem desempenhar um papel crucial nesta expansão da rede de interação dos genes. Um número substancial destas moléculas surgiu no momento da aquisição de lamelócitos, elas são expressas no momento certo na resposta imunológica, e mostram fortes assinaturas de seleção positiva (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
Também dentro de uma única espécie, D. melanogaster, as respostas imunológicas mostram grande variação genética. As populações de campo coletadas de toda a Europa mostram diferenças substanciais na capacidade de encapsular com sucesso ovos parasitoides (Kraaijeveld e van Alphen, 1995; Kraaijeveld e Godfray, 1999; Gerritsma et al., 2013). Aparentemente, os custos e benefícios de uma forte defesa imunológica diferem geograficamente, levando à modulação e diferenciação das redes genéticas co-adaptadas. Isto também se reflectiu na resposta hemocítica após o ataque de parasitoides. As linhas de campo variaram consideravelmente nos números absolutos e relativos dos diferentes hemócitos que produziram em resposta ao ataque de parasitoides, mesmo entre as linhas que tiveram grande sucesso no encapsulamento (Gerritsma et al., 2013). Isto enfatiza novamente que o fundo genético de uma população e as pressões combinadas de seleção local levam a respostas evolutivas alternativas. A comparação dos genomas de indivíduos resistentes e susceptíveis de várias populações pode revelar a variação adaptativa na arquitetura genética desta característica.
Para mapear as mudanças no genoma durante a evolução do aumento da resistência, realizamos a evolução experimental para a resistência aos parasitóides. Em laboratório, expusemos uma grande população de parasitóides. Apenas as larvas que conseguiram sobreviver ao ataque de parasitoides foram autorizadas a contribuir para a geração seguinte. Com esta abordagem, aumentamos o nível de resistência de 20 para 4% das larvas que sobreviveram ao ataque de parasitoides após apenas cinco gerações de seleção. Quando medimos as mudanças na expressão gênica nas populações selecionadas, em comparação com a expressão gênica nas linhas de controle, mesmo antes do ataque parasitoide, encontramos várias centenas de genes que foram ligeiramente regulados de forma diferente (Wertheim et al., 2011). As mudanças envolveram principalmente genes que não foram diferentemente expressos durante a resposta imune, indicando que as mudanças evolutivas não pré-ativaram a resposta imune em antecipação ao ataque de parasitoides, mas modularam a via de desenvolvimento canônico, o que (também) levou a um aumento de suas capacidades defensivas. Repetimos esta experiência, e depois sequenciamos os genomas das populações seleccionadas e controladas. Nos genomas das linhas que evoluíram com o aumento da resistência, encontramos assinaturas de seleção em múltiplas regiões estreitamente definidas do genoma (Jalvingh et al., 2014). Algumas dessas regiões também se sobrepuseram às regiões que mostraram expressão alterada após a seleção para maior resistência (Wertheim et al., 2011; Jalvingh et al., 2014). Assim, uma varredura seletiva rápida e forte sobre uma característica complexa como a imunidade ainda pode afetar múltiplas, mas altamente localizadas, regiões genômicas.
Desafios Futuros
Como vamos reconciliar as mudanças evolutivas de longo prazo, como a aquisição de novos genes em redes de interação genética, e as mudanças evolutivas de curto prazo, como as variantes de seqüência que podem ser varridas através de uma população? A chave para isso é (i) reconstruir a rede de interação genética subjacente aos traços complexos, e (ii) caracterizar o papel da variação genética dentro dessas redes. As redes genéticas podem expandir-se com novos genes através, por exemplo, de duplicações, interligar-se com outras redes ou módulos, e pequenas variações de sequência podem modular a actividade e a topologia das redes. Se conseguirmos decompor as redes genéticas e avaliar o papel das variantes genéticas no contexto de uma rede, isso nos permitirá eventualmente determinar como a variação genética é traduzida em variação fenotípica. Isto também melhorará nossa compreensão da base molecular de doenças humanas complexas e da evolução da imunidade inata e adquirida (Cooper e Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay e Moore, 2014; Sironi et al., 2015).
As abordagens biológicas dos sistemas serão inestimáveis para desvendar as complexas redes de interação genética. Lá, modelos matemáticos são desenvolvidos para descrever os mecanismos moleculares subjacentes a uma característica e para prever a dinâmica de grupos de componentes de interação da rede. Os modelos são baseados na genética molecular e em dados genómicos. Atualmente, a biologia de sistemas é aplicada principalmente a traços específicos em organismos unicelulares, e isso é considerado o limite do que pode ser alcançado (Papp et al., 2011). É provável, no entanto, que este modelo possa não ser representativo da evolução em organismos multicelulares sexualmente reprodutores. Ao invés de esperar que os modelos simplificados reflitam com precisão pequenas sub-redes, precisamos desenvolver e refinar nossos métodos para utilizar e quantificar as propriedades emergentes a partir da vasta quantidade de dados genômicos. Podemos inferir redes de interação de genes a partir de matrizes de correlação ou co-expressão proteína-proteína ou transcript (Shannon et al., 2003; Langfelder e Horvath, 2008), e fundi-las com análises de variantes naturais (Nuzhdin et al., 2012). Devemos continuar a desenvolver estes métodos e abordagens alternativas para explorar plenamente as nossas medições em dados genómicos e converter estas medições quantitativas em análises em rede. Enquanto os dados genómicos em si mesmos não vão fornecer a resposta completa para o que determina a capacidade adaptativa da vida, permitem-nos quantificar e observar o que acontece a nível molecular durante a evolução. Quando combinamos e integramos isto com a heterogeneidade ambiental como motor das adaptações, podemos ser capazes de revelar os complexos mecanismos moleculares de adaptação e evolução.
Conflict of Interest Statement
O autor declara que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Acknowledgments
Eu agradeço a Kirsten Jalvingh, Laura Salazar-Jaramillo, e Sylvia Gerritsma pelas valiosas discussões. A BW foi apoiada por financiamento da Organização Holandesa de Pesquisa Científica (NWO; grant 864.08.008).
Aggarwal, K., e Silverman, N. (2008). Regulação positiva e negativa da resposta imunológica de Drosophila. BMB Rep. 41, 267-277. doi: 10.5483/BMBRBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Genética de sistemas de características complexas em Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., e Anderson, K. V. (2004). Drosophila: a genética do reconhecimento e resposta imunológica inata. Annu. Rev. Imunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evolução da imunidade inata: pistas de invertebrados via peixes para mamíferos. Frente. Imunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., e Fitzgerald, K. A. (2014). Regulação pós-transcrítica da expressão gênica na imunidade inata. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., e Alder, M. N. (2006). A evolução dos sistemas imunitários adaptativos. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biologia, molecular e organísmica. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Evolução dos genes e genomas na filogenia de Drosophila. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., and Mackay, T. F. C. (2009). Uma rede transcripcional associada à variação natural do comportamento agressivo de Drosophila. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., e Doury, G. (2006). A mosca Drosophila subobscura: um caso natural de deficiência de imunidade inata. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., e Banerjee, U. (2003). Mais espessos que o sangue: mecanismos conservados em Drosophila e hematopoiese de vertebrados. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D., e Eisen, M. B. (2004). A variação genética da população na expressão gênica está associada à variação fenotípica em Saccharomyces cerevisiae. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., and Allemand, R. (2009). Ecologia e evolução da história de vida dos parasitóides Drosophila frugívoros. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Programas de expressão genómica na resposta das células de levedura às mudanças ambientais. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., e Wertheim, B. (2013). A variação natural dos hemócitos diferenciados está relacionada com a resistência aos parasitóides na Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., and Eslin, P. (2012). Caracterização estrutural e funcional de pseudopodócitos, uma célula imune desgrenhada produzida por duas espécies de Drosophila do grupo das obscuras. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). A evolução experimental, análise genética e re-sequenciamento genético revelam a mutação conferindo resistência à artemisinina em uma linhagem isogênica de parasitas da malária. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., and Anderson, P. (2013). Redes reguladoras pós-transcritivas em imunidade. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., e Wertheim, B. (2014). Alterações genómicas em evolução rápida: selecção para resistência a parasitoides. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). A base genómica da evolução adaptativa em sticklebacks triplos. Nature 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Unraveling adaptive evolution: how a single point mutation affects the protein coregulation network. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and Godfray, H. C. J. (1999). Padrões geográficos na evolução da resistência e da virulência em Drosophila e seus parasitóides. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., e Godfray, H. C. J. (2002). Costs of resistance in insect-parasite and insect-parasitoid interactions. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., e van Alphen, J. J. M. (1995). Geographical variation in encapsulation ability of Drosophila melanogaster larvae and evidence for parasitoid-specific components. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., e Horvath, S. (2008). WGCNA: um pacote R para análise de rede de correlação ponderada. BMC Bioinformática 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., and Strand, M. R. (2002). Os hemócitos dos insetos e seu papel na imunidade. Biochem do inseto. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., e Little, T. J. (2009). Imunidade em um mundo variável. Filosofia. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., e Schneider, D. S. (2014). A genética da imunidade. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Genótipo para fenótipo: lições de organismos modelo para a genética humana. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., and Hoffmann, J. (2007). A defesa anfitriã da Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., e O’Neill, L. A. J. (2005). Regulação negativa das respostas imunitárias mediadas por receptores. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). Evolução adaptativa de múltiplos traços através de múltiplas mutações em um único gene. Science 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., and Moore, J. H. (2014). Por que a epistasia é importante para enfrentar a complexa genética das doenças humanas. Genoma Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). Finding the missing heritability of complex diseases. Nature 461, 747-753. doi: 10.1038/natureza08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Os hemócitos gigantes multinucleados são células efetoras das respostas imunes mediadas por células de Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., and Williams, S. M. (eds). (2015). Epistasia. Nova York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., e Plasterk, R. (2003). A adaptação do genoma: o poder das abordagens genéticas. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., and Stubbs, L. (2009). As diferenças nos padrões de expressão gênica humana e dos chimpanzés definem uma rede evolutiva de fatores de transcrição no cérebro. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., e McIntyre, L. M. (2012). Mapeamento genótipo-fenótipo em um mundo pós-GWAS. Tendências Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J., Kim, K.-W., e Jiggins, F. M. (2009). Quantificando a evolução adaptativa no sistema imunológico de Drosophila. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., e Pál, C. (2011). Abordagens de sistemas-biologia para prever a evolução genómica. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., e Pittendrigh, B. R. (2004). Genome-wide transcription profile of field- and laboratory-selected dichlorodiphenyltrichloroethane (DDT)-resistant Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistasia – o papel essencial das interações genéticas na estrutura e evolução dos sistemas genéticos. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., e Siva-Jothy, M. T. (2003). Imunologia Ecológica Invertebrada. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., and Clark, A. G. (2007). Dynamic evolution of the innate immune system in Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., and Wertheim, B. (2014). Evolução de uma resposta imune celular em Drosophila: uma análise fenotípica e genómica comparativa. Biol do Genoma. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). As redes moleculares como sensores e condutores de doenças humanas comuns. Nature 461, 218-223. doi: 10.1038/natureza08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., e Clark, A. G. (2007). Estratégias contrastantes de infecção em parasitóides de vespas generalistas e especializadas da Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variação na defesa imunológica como uma questão de ecologia evolutiva. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Ecologia evolutiva das defesas imunitárias dos insectos. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: um ambiente de software para modelos integrados de redes de interação biomolecular. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., and Maniatis, T. (2001). NF-κB sinalização de vias de sinalização em imunidade inata de mamíferos e insetos. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., e Clerici, M. (2015). Perspectivas evolutivas sobre as interacções hospedeiro-patógeno a partir de dados de sequência de mamíferos. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Adaptation genomics: the next generation. Tendências Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). A sequência genómica do bacalhau do Atlântico revela um sistema imunitário único. Nature 477, 207-210. doi: 10.1038/natureza10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Cem anos de pleiotropia: uma retrospectiva. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., and Wheat, C. W. (2010). Integrando abordagens evolutivas e funcionais para inferir a adaptação em loci específicos. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). A diversidade molecular da convergência adaptativa. Science 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., e Tarone, A. M. (2011). A ressequenciação populacional de populações experimentalmente evoluídas revela a base genética da variação do tamanho do corpo em Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., e Altenberg, L. (1996). Adaptações complexas e a evolução da evolutividade. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., and Godfray, H. C. (2011). Genómica funcional da evolução do aumento da resistência ao parasitismo em Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Expressão genética em resposta ao ataque de parasitoides em Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., e Aderem, A. (2014). Análise da imunidade inata a nível de sistemas. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar