Fronteras de la Genética
Introducción
Muchos rasgos están regulados y coordinados por múltiples genes y condiciones ambientales. En la naturaleza, estos rasgos evolucionan continuamente bajo presiones de selección locales y procesos neutros, lo que da lugar a una rica diversidad de variedades fenotípicas y estrategias de adaptación al medio ambiente. Ya Dobzhansky (1964) describió la rica diversidad del mundo viviente como el resultado de la diversidad genética, la heterogeneidad ambiental y las adaptaciones que evolucionan en la interacción entre la biología molecular y la organísmica. Los mecanismos moleculares de la evolución han sido especialmente difíciles de caracterizar. En primer lugar, hay que vincular el genotipo con el fenotipo. Este vínculo consiste en extensas e intrincadas redes de interacción genética (Ayroles et al., 2009; Lehner, 2013). En segundo lugar, necesitamos identificar los cambios moleculares responsables de las adaptaciones fenotípicas. Esto depende de la elucidación de la variación genética y los cambios genéticos que pueden ocurrir en cualquier parte de las redes de interacción de genes (por ejemplo, Edwards et al., 2009).
La manifestación de la variación genética en las redes de genes es muy compleja. Los cambios en genes individuales pueden afectar a la actividad e incluso a la topología de toda la red genética (por ejemplo, Knight et al., 2006). Los genes y las redes de interacción génica suelen ser pleiotrópicos y regulan varios rasgos y procesos, lo que implica que los cambios en un solo gen pueden manifestarse en varios rasgos (Stearns, 2010). A la inversa, las variaciones alélicas en muchos genes pueden contribuir a la variación de un rasgo fenotípico concreto (Manolio et al., 2009). Además, la epistasis es omnipresente, lo que implica que las variaciones alélicas en múltiples loci pueden afectar a los efectos fenotípicos de cada uno (Phillips, 2008; Mackay y Moore, 2014; Moore y Williams, 2015). Estas consideraciones sobre la base genética de la evolución no son nuevas, y se han estudiado durante varias décadas (por ejemplo, Wagner y Altenberg, 1996). La genética directa e inversa ha tenido mucho éxito a la hora de dilucidar las funciones de genes individuales o mutaciones para un rasgo concreto (Nagy et al., 2003). Sin embargo, estas técnicas son limitadas a la hora de estudiar la complejidad de las redes de interacción molecular que subyacen a un fenotipo, o los mecanismos moleculares de la evolución de rasgos complejos.
Los avances en la tecnología genómica han supuesto un gran impulso para nuestra capacidad de estudiar las complejidades genéticas de los rasgos fenotípicos y su evolución (Stapley et al., 2010). La combinación de estas técnicas con los enfoques clásicos de la genética nos permite evaluar la funcionalidad de la variación genética para los rasgos fenotípicos (Storz y Wheat, 2010). Los primeros estudios genómicos en varios organismos modelo destacaron que las adaptaciones evolutivas, incluso para condiciones ambientales específicas, suelen gobernar muchos genes o loci, así como la regulación dinámica de los patrones de expresión génica (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). En la década siguiente, muchos estudios utilizaron la genómica para identificar los genes y las proteínas que contribuían a determinados rasgos e interacciones ecológicas. Al principio, los costes y el tiempo necesarios para secuenciar un solo genoma eran todavía muy restrictivos. Sin embargo, la secuenciación de próxima generación ha permitido secuenciar los genomas de muchas más especies y de muchos más individuos por especie. Se trata de un recurso formidable para estudiar la evolución, ya que nos permite, por primera vez, cartografiar los cambios en todo el genoma durante la evolución.
La tecnología genómica tiene un enorme potencial para mejorar nuestros conocimientos sobre los procesos evolutivos. Se han aplicado enfoques comparativos para mapear los cambios en las secuencias del genoma o en las redes de interacción de los genes en largas escalas de tiempo evolutivo (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Los enfoques de selección experimental o evolución experimental, seguidos de la transcriptómica o la secuenciación del genoma, se han utilizado para mapear los cambios evolutivos a escalas de tiempo mucho más cortas (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Estos estudios mostraron, por ejemplo, cómo las duplicaciones de genes, las mutaciones y la fuerte divergencia de secuencias en un pequeño subconjunto de genes pueden tener un profundo impacto en la actividad transcripcional de grandes redes de interacción de genes y en múltiples rasgos fenotípicos.
En esta perspectiva, presento los descubrimientos y desarrollos recientes sobre las bases genómicas de la evolución, utilizando la inmunidad evolutiva como caso de estudio. La inmunidad es un rasgo que evoluciona rápidamente, por lo que es susceptible de estudiar las bases genómicas de los procesos evolutivos (Obbard et al., 2009; Sironi et al., 2015). Además, las redes moleculares en la inmunidad han sido bastante bien caracterizadas debido a su importancia para la salud humana (Schadt, 2009; Lazzaro y Schneider, 2014; Zak et al., 2014). En primer lugar, resumo muy brevemente nuestros conocimientos actuales sobre las redes moleculares de las respuestas inmunitarias innatas y los procesos de selección que actúan sobre las respuestas inmunitarias. A continuación, describo los cambios genómicos, asociados con la ganancia, la pérdida y la modulación de aspectos particulares de las respuestas inmunes en Drosophila. Finalmente, propongo futuras direcciones para estudiar la arquitectura genética de rasgos complejos y procesos evolutivos.
Respuestas inmunitarias
El sistema inmunitario consiste en una combinación de procesos fisiológicos que actúan conjuntamente en la defensa contra patógenos y parásitos. La inmunidad innata es un rasgo antiguo que se encuentra en todos los organismos multicelulares, mientras que los vertebrados también poseen inmunidad adquirida. Ambos sistemas inmunitarios combinan componentes celulares y humorales: el componente celular comprende células especializadas que proporcionan una función protectora. Esto incluye, clases de células sanguíneas para la fagocitosis de microbios, la encapsulación de cuerpos extraños más grandes o el reconocimiento de antígenos (en la inmunidad adquirida), y el revestimiento del intestino con células epiteliales que forman una barrera física y pueden secretar compuestos defensivos. El componente humoral consiste en la liberación de factores extracelulares que combaten los patógenos invasores, a menudo desde tejidos o poblaciones celulares especializados. Esto incluye la liberación de péptidos antimicrobianos (AMP) por parte del hígado (o el cuerpo graso en los invertebrados) y las células epiteliales del intestino, las especies reactivas del oxígeno en las células fagocíticas y epiteliales, y los anticuerpos de los glóbulos blancos (en la inmunidad adquirida; Lemaitre y Hoffmann, 2007; Buchmann, 2014).
Complejas redes de interacciones moleculares coordinan las respuestas inmunitarias (Figura 1A). Las mismas vías son fundamentales para las respuestas inmunitarias desde los invertebrados hasta los vertebrados, lo que implica una fuerte conservación de los elementos centrales de las redes moleculares en la inmunidad (Silverman y Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). Una variedad de moléculas receptoras pueden reconocer a los patógenos o parásitos, por ejemplo, basándose en patrones moleculares asociados a los patógenos (por ejemplo, lipopolisacáridos específicos de las membranas bacterianas). Una vez que los receptores se activan, inducen vías de transducción de señales específicas, como las vías Toll, Imd y Jak/Stat (revisadas en Lemaitre y Hoffmann, 2007; Buchmann, 2014). Estas vías consisten en proteasas, quinasas, citoquinas y otras proteínas que finalmente activan factores de transcripción y cofactores. La inducción de estos factores de transcripción da lugar a la producción de moléculas efectoras humorales (por ejemplo, AMPs) y puede inducir la proliferación y diferenciación de las células implicadas en la inmunidad. La producción de diferentes clases de células sanguíneas es un aspecto destacado del componente celular de la respuesta inmunitaria, tanto en la inmunidad innata como en la adquirida. En estas células sanguíneas también se regulan las cascadas de transducción de señales para inducir las propiedades de las células y las proteínas que efectúan la eliminación del parásito. Para regular la fuerza, la especificidad, el momento y la duración de las respuestas inmunitarias, las redes moleculares son moduladas por citoquinas, proteasas y la interacción con otras vías de señalización (Liew et al., 2005; Aggarwal y Silverman, 2008). Esto también incluye diversas redes de regulación postranscripcional (Ivanov y Anderson, 2013; Carpenter et al., 2014).
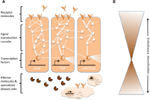
Figura 1. Representación esquemática de las redes genéticas en la inmunidad. (A) Varias redes interconectadas coordinan las respuestas a un desafío inmunológico. Estas redes consisten en proteínas (representadas por círculos) que interactúan entre sí en una cascada de transducción de señales para regular la expresión de factores de transcripción (representados por hexágonos). La activación de las vías centrales de transducción de señales (por ejemplo, IMD, Toll o Jak/Stat, indicadas por líneas gruesas entre las proteínas) da lugar a la producción de moléculas efectoras, como péptidos antimicrobianos (representados por símbolos en forma de tarta) y a la proliferación y diferenciación de células especializadas (sanguíneas) (figuras en forma de nube). Las moléculas receptoras extracelulares y unidas a la membrana (figuras en forma de luna) inducen las vías. La actividad puede ser modulada además por muchas otras proteínas que interactúan con las vías y se cruzan con otras vías y redes genéticas (indicadas por las líneas finas entre las proteínas). (B) Los componentes centrales de las redes genéticas en la inmunidad, por ejemplo, los factores de transcripción y las proteínas en contacto directo con estos factores de transcripción, suelen estar muy conservados entre los distintos filos. La diversificación evolutiva se encuentra más ampliamente hacia la periferia de las redes.
Los impulsores de los cambios evolutivos en las respuestas inmunitarias son los efectos combinados de los altos costes de aptitud de la infección en los hospedadores, los costes de la inmunidad, la rica diversidad de patógenos y parásitos que amenazan a los hospedadores, y las dinámicas carreras armamentísticas coevolutivas entre los hospedadores y los patógenos (Schmid-Hempel, 2003). Cualquiera de estos aspectos, o todos ellos, pueden operar en el entorno local del hospedador y provocar fuertes presiones de selección. El tipo de selección, sin embargo, varía desde la direccional hasta la purificadora o la equilibradora, dependiendo de los costes y beneficios que la población del huésped experimenta en su entorno local. Por ejemplo, un patógeno muy virulento que invade una comunidad local puede provocar un barrido selectivo o una selección direccional para determinados alelos de resistencia, ya que sólo los hospedadores con estos alelos pueden contribuir a las siguientes generaciones. Por otro lado, una comunidad de patógenos diversa o en coevolución puede impulsar una selección dependiente de la frecuencia o de equilibrio, favoreciendo el mantenimiento de la variación genética. La evolución de las respuestas inmunitarias refleja tanto esta dinámica coevolutiva con el parásito, como los costes fisiológicos y ecológicos del sistema inmunitario (Kraaijeveld et al., 2002; Rolff y Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro y Little, 2009).
Base genómica del cambio evolutivo en la inmunidad de Drosophila
Mientras que la maquinaria central de las respuestas inmunitarias está fuertemente conservada, varios componentes de las redes moleculares extendidas pueden evolucionar rápidamente o diversificarse (Figura 1B). En Drosophila, se ha reportado un rápido cambio evolutivo para los receptores y los efectores de la respuesta inmune (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Estas moléculas operan en la interfaz entre el hospedador y el patógeno, y por lo tanto son cruciales para el reconocimiento por parte del hospedador de un organismo invasor, y para mediar la focalización y los efectos antagónicos de la respuesta inmune sobre el patógeno. Al mismo tiempo, el parásito está bajo selección para pasar desapercibido, para evitar o mitigar los efectos antagónicos de la respuesta inmune. Por lo tanto, se espera una dinámica de Reina Roja para estas moléculas en la interfaz, y las que modulan las respuestas inmunitarias. Cada una de las partes trata de imponerse en la carrera armamentística antagónica, impulsando recíprocamente alteraciones en las redes genéticas de las partes. La diversificación en las moléculas receptoras, moduladoras y efectoras se lleva a cabo principalmente mediante duplicaciones de genes y rápidos cambios de secuencia (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Hemos estado estudiando la respuesta inmune de Drosophila contra las avispas parasitoides como un sistema modelo para entender las bases genómicas de los procesos evolutivos. Las larvas de Drosophila son huéspedes de una variedad de especies de parasitoides que ponen un huevo en estas larvas (Fleury et al., 2009). Una vez que el huevo del parasitoide eclosiona (∼2-4 días después del ataque del parasitoide, dependiendo de la especie del parasitoide, y de la temperatura), la larva del parasitoide comienza a alimentarse del huésped y lo mata. Algunas especies de Drosophila tienen un mecanismo de defensa contra los parasitoides a través de una respuesta inmune innata, llamada encapsulación melanótica. Esta respuesta inmunitaria consta de componentes celulares y humorales que actúan conjuntamente para secuestrar y matar el huevo del parasitoide. El ataque del parasitoide desencadena vías de transducción de señales inmunitarias que inducen (i) la proliferación y diferenciación de dos clases de hemocitos (es decir, células sanguíneas de los insectos) que se adhieren al huevo del parasitoide y entre sí, y (ii) la deposición de melanina sobre el huevo del parasitoide y la cápsula celular que lo rodea (Lemaitre y Hoffmann, 2007). El hospedador tiene que completar el encapsulamiento y la melanización antes de que el huevo del parasitoide eclosione para sobrevivir a la infestación del parasitoide.
Antes de la era de la genómica, se habían identificado varios genes implicados en la respuesta inmunitaria contra las avispas parasitoides. Se habían identificado las vías Toll y Jak/Stat como componentes centrales de la proliferación y diferenciación de hemocitos, y la vía de la profenoloxidasa para la melanización (revisado en Brennan y Anderson, 2004). A continuación, se llevaron a cabo dos estudios de microarrays para identificar otros genes potencialmente implicados en la encapsulación melanótica tras el ataque del parasitoide. Este enfoque puso de manifiesto muchos genes adicionales que no se habían asociado previamente a la respuesta inmunitaria contra el parasitoide, y reveló su momento de acción (Wertheim et al., 2005; Schlenke et al., 2007). Los estudios revelaron varios grupos coordinados y funcionalmente coherentes de genes que fueron temporalmente regulados al alza o a la baja durante parte de la respuesta inmunitaria (Wertheim et al., 2005). Curiosamente, se demostró que los mecanismos de virulencia de dos especies de parasitoides diferían en la forma en que interferían en la red genética de las respuestas de los hospedadores: una especie eliminaba la activación inicial de toda la red, mientras que otra especie se dirigía al último paso de la cascada (Schlenke et al., 2007).
Las especies de Drosophila difieren en gran medida en la inmunidad contra los parasitoides. Algunas especies son completamente susceptibles a los parasitoides y esto fue reportado como una inmunodeficiencia (Eslin y Doury, 2006). Sin embargo, una inspección más detallada reveló que la resistencia a los parasitoides no es comúnmente compartida entre todas las especies de Drosophila, sino que está restringida a unos pocos clados. Demostramos que en uno de esos clados, el subgrupo melanogaster, la ganancia evolutiva de la resistencia a los parasitoides estaba asociada a la ganancia de un nuevo tipo de célula sanguínea, los lamelocitos, que también está restringida al mismo clado (Salazar-Jaramillo et al., 2014). Algunas especies de Drosophila fuera del subgrupo melanogaster también pueden encapsular huevos de parasitoides, pero parecen haber evolucionado diferentes tipos de células sanguíneas para la respuesta de encapsulación (Havard et al., 2012; Márkus et al., 2015). La respuesta inmunitaria contra los parasitoides ha evolucionado de forma independiente en varios taxones de insectos, a menudo con mecanismos y tipos de células sanguíneas ligeramente diferentes (Lavine y Strand, 2002). Así, la evolución del sistema inmunitario innato incluye la adición de nuevos componentes o «módulos». Esto plantea la cuestión de cómo cambia el genoma durante la adquisición de un nuevo módulo.
La genómica comparativa reveló que, a pesar de la ganancia de un nuevo tipo de célula sanguínea en el subgrupo melanogaster, los genes que se conocen para la diferenciación de los lamelocitos se conservan en gran medida en toda la filogenia. También las especies que no producen lamelocitos en respuesta al ataque de los parasitoides poseen estos genes necesarios para la diferenciación de los lamelocitos. Además, estos genes muestran poca divergencia o firmas de selección, mientras que eso sería lo esperado para los genes que obtuvieron una función novedosa. Esto indica que las vías de transducción de señales existentes para la diferenciación de los hemocitos están siendo moduladas por la red de interacción genética circundante para producir un nuevo tipo de célula sanguínea en el subgrupo melanogaster. Esta cooptación de la vía central de proliferación de hemocitos existente se consigue probablemente añadiendo otros o nuevos componentes a la red de interacción génica (Salazar-Jaramillo et al., 2014). Identificamos varios genes nuevos que surgieron alrededor del momento de la adquisición de lamelocitos y que se expresan diferencialmente durante la respuesta inmune contra los parasitoides, incluyendo moléculas receptoras y proteasas de tipo serina (Salazar-Jaramillo et al., 2014). Nuestra hipótesis es que especialmente las endopeptidasas de tipo serina pueden desempeñar un papel crucial en esta expansión de la red de interacción de genes. Un número sustancial de estas moléculas surgió en el momento de la adquisición de los lamelitos, se expresan en el momento adecuado en la respuesta inmune, y muestran fuertes firmas de selección positiva (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
También dentro de una sola especie, D. melanogaster, las respuestas inmunes muestran una gran variación genética. Las poblaciones de campo recogidas en toda Europa muestran diferencias sustanciales en la capacidad de encapsular con éxito los huevos de parasitoides (Kraaijeveld y van Alphen, 1995; Kraaijeveld y Godfray, 1999; Gerritsma et al., 2013). Aparentemente, los costes y beneficios de una fuerte defensa inmunitaria difieren geográficamente, lo que lleva a la modulación y diferenciación de las redes genéticas coadaptadas. Esto también se reflejó en la respuesta hemocítica tras el ataque del parasitoide. Las líneas de campo variaron considerablemente en los números absolutos y relativos de los diferentes hemocitos que produjeron en respuesta al ataque de los parasitoides, incluso entre las líneas que tuvieron mucho éxito en la encapsulación (Gerritsma et al., 2013). Esto vuelve a enfatizar que los antecedentes genéticos de una población y las presiones de selección locales combinadas conducen a respuestas evolutivas alternativas. La comparación de los genomas de individuos resistentes y susceptibles de varias poblaciones puede revelar la variación adaptativa en la arquitectura genética de este rasgo.
Para mapear los cambios en el genoma durante la evolución del aumento de la resistencia, llevamos a cabo la evolución experimental para la resistencia de los parasitoides. En el laboratorio, expusimos una gran población de crías a los parasitoides. Sólo las larvas que lograron sobrevivir al ataque de los parasitoides pudieron contribuir a la siguiente generación. Con este enfoque, aumentamos el nivel de resistencia del 20 al ∼50% de las larvas que sobrevivían al ataque de los parasitoides tras sólo cinco generaciones de selección. Cuando medimos los cambios en la expresión de los genes en las poblaciones seleccionadas, en comparación con la expresión de los genes en las líneas de control, incluso antes del ataque del parasitoide, encontramos varios cientos de genes que estaban ligeramente regulados de forma diferencial (Wertheim et al., 2011). Los cambios implicaban sobre todo genes que no se expresaban diferencialmente durante la respuesta inmunitaria, lo que indica que los cambios evolutivos no preactivaron la respuesta inmunitaria en previsión del ataque del parasitoide, sino que moduló la vía de desarrollo canónica, lo que (también) condujo a un aumento de sus capacidades defensivas. Repetimos este experimento, y luego secuenciamos los genomas de las poblaciones seleccionadas y de control. En los genomas de las líneas que desarrollaron una mayor resistencia, encontramos firmas de selección en múltiples regiones del genoma estrechamente definidas (Jalvingh et al., 2014). Algunas de estas regiones también se superponen con las regiones que mostraron cambios en la expresión después de la selección para el aumento de la resistencia (Wertheim et al., 2011; Jalvingh et al., 2014). Así, un barrido selectivo rápido y fuerte sobre un rasgo complejo como la inmunidad puede seguir afectando a regiones genómicas múltiples, pero muy localizadas.
Desafíos futuros
¿Cómo vamos a conciliar los cambios evolutivos a largo plazo, como la adquisición de nuevos genes en las redes de interacción génica, y los cambios evolutivos a corto plazo, como las variantes de secuencia que pueden ser barridas por una población? La clave para ello es (i) reconstruir la red de interacción génica que subyace a los rasgos complejos, y (ii) caracterizar el papel de la variación genética dentro de estas redes. Las redes genéticas pueden ampliarse con nuevos genes mediante, por ejemplo, duplicaciones, interconectarse con otras redes o módulos, y pequeñas variaciones de secuencia pueden modular la actividad y la topología de las redes. Si podemos descomponer las redes genéticas y evaluar el papel de las variantes genéticas en un contexto de red, esto nos permitirá finalmente determinar cómo se traduce la variación genética en variación fenotípica. Esto también mejorará nuestra comprensión de las bases moleculares de las complejas enfermedades humanas y de la evolución de la inmunidad innata y adquirida (Cooper y Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay y Moore, 2014; Sironi et al., 2015).
Los enfoques biológicos de sistemas serán muy valiosos para desentrañar las complejas redes de interacción genética. En ellos, se desarrollan modelos matemáticos para describir los mecanismos moleculares que subyacen a un rasgo y para predecir la dinámica de los grupos de componentes que interactúan en la red. Los modelos se basan en datos de genética molecular y genómica. En la actualidad, la biología de sistemas se aplica sobre todo a rasgos específicos de organismos unicelulares, lo que se considera el límite de lo que se puede conseguir (Papp et al., 2011). Sin embargo, es probable que este modelo no sea representativo de la evolución en los organismos multicelulares que se reproducen sexualmente. En lugar de esperar a que los modelos simplificados reflejen con precisión las pequeñas subredes, debemos desarrollar y perfeccionar nuestros métodos para utilizar y cuantificar las propiedades emergentes a partir de la gran cantidad de datos genómicos. Podemos inferir redes de interacción génica a partir de la correlación proteína-proteína o transcripción o matrices de coexpresión (Shannon et al., 2003; Langfelder y Horvath, 2008), y fusionarlas con análisis de variantes naturales (Nuzhdin et al., 2012). Deberíamos seguir desarrollando estos métodos y enfoques alternativos para explotar plenamente nuestras mediciones en los datos genómicos, y convertir estas mediciones cuantitativas en análisis de redes. Aunque los datos genómicos en sí mismos no van a proporcionar la respuesta completa a lo que determina la capacidad de adaptación de la vida, nos permiten cuantificar y observar lo que ocurre a nivel molecular durante la evolución. Cuando combinemos e integremos esto con la heterogeneidad ambiental como impulsora de las adaptaciones, podremos revelar los complejos mecanismos moleculares de la adaptación y la evolución.
Declaración de conflicto de intereses
El autor declara que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
Agradezco a Kirsten Jalvingh, Laura Salazar-Jaramillo y Sylvia Gerritsma por sus valiosas discusiones. BW fue apoyado por la financiación de la Organización Holandesa para la Investigación Científica (NWO; subvención 864.08.008).
Aggarwal, K., y Silverman, N. (2008). Regulación positiva y negativa de la respuesta inmune de Drosophila. BMB Rep. 41, 267-277. doi: 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Systems genetics of complex traits in Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., y Anderson, K. V. (2004). Drosophila: la genética del reconocimiento y la respuesta inmune innata. Annu. Rev. Immunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evolución de la inmunidad innata: pistas desde los invertebrados hasta los mamíferos, pasando por los peces. Front. Immunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., and Fitzgerald, K. A. (2014). Regulación post-transcripcional de la expresión génica en la inmunidad innata. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., y Alder, M. N. (2006). La evolución de los sistemas inmunitarios adaptativos. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biología molecular y organísmica. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Evolución de genes y genomas en la filogenia de Drosophila. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., y Mackay, T. F. C. (2009). Una red transcripcional asociada a la variación natural en el comportamiento agresivo de Drosophila. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., y Doury, G. (2006). La mosca Drosophila subobscura: un caso natural de deficiencia de inmunidad innata. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., y Banerjee, U. (2003). Thicker than blood: conserved mechanisms in Drosophila and vertebrate hematopoiesis. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D., y Eisen, M. B. (2004). Población variación genética en la expresión de genes se asocia con la variación fenotípica en Saccharomyces cerevisiae. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., y Allemand, R. (2009). Ecology and life history evolution of frugivorous Drosophila parasitoids. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Programas de expresión genómica en la respuesta de las células de levadura a los cambios ambientales. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., and Wertheim, B. (2013). La variación natural en los hemocitos diferenciados está relacionada con la resistencia a los parasitoides en Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., y Eslin, P. (2012). Caracterización estructural y funcional del pseudopodocito, una célula inmune peluda producida por dos especies de Drosophila del grupo obscura. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). Experimental evolution, genetic analysis and genome re-sequencing reveal the mutation conferring artemisinin resistance in an isogenic lineage of malaria parasites. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., y Anderson, P. (2013). Redes reguladoras post-transcripcionales en la inmunidad. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., and Wertheim, B. (2014). Cambios genómicos bajo una rápida evolución: selección para la resistencia a los parasitoides. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). The genomic basis of adaptive evolution in threespine sticklebacks. Nature 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Unraveling adaptive evolution: how a single point mutation affects the protein coregulation network. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., y Godfray, H. C. J. (1999). Patrones geográficos en la evolución de la resistencia y la virulencia en Drosophila y sus parasitoides. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., y Godfray, H. C. J. (2002). Costs of resistance in insect-parasite and insect-parasitoid interactions. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and van Alphen, J. J. M. (1995). Geographical variation in encapsulation ability of Drosophila melanogaster larvae and evidence for parasitoid-specific components. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., and Horvath, S. (2008). WGCNA: un paquete R para el análisis de redes de correlación ponderada. BMC Bioinformatics 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., and Strand, M. R. (2002). Insect hemocytes and their role in immunity. Insect Biochem. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Little, T. J. (2009). Inmunidad en un mundo variable. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., y Schneider, D. S. (2014). La genética de la inmunidad. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Genotipo a fenotipo: lecciones de los organismos modelo para la genética humana. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., y Hoffmann, J. (2007). La defensa del huésped de Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., y O’Neill, L. A. J. (2005). Regulación negativa de las respuestas inmunitarias mediadas por el receptor Toll-like. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). Adaptive evolution of multiple traits through multiple mutations at a single gene. Science 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., y Moore, J. H. (2014). Por qué la epistasis es importante para abordar la genética de las enfermedades humanas complejas. Genome Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). Finding the missing heritability of complex diseases. Nature 461, 747-753. doi: 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Multinucleated giant hemocytes are effector cells in cell-mediated immune responses of Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., y Williams, S. M. (eds). (2015). Epistasis. New York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., y Plasterk, R. (2003). Adaptar el genoma: el poder de los enfoques genéticos. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., y Stubbs, L. (2009). Las diferencias en los patrones de expresión genética de humanos y chimpancés definen una red evolutiva de factores de transcripción en el cerebro. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., and McIntyre, L. M. (2012). Mapeo genotipo-fenotipo en un mundo post-GWAS. Trends Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J., Kim, K.-W., and Jiggins, F. M. (2009). Cuantificación de la evolución adaptativa en el sistema inmunológico de Drosophila. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., y Pál, C. (2011). Enfoques de biología de sistemas para predecir la evolución genómica. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., and Pittendrigh, B. R. (2004). Genome-wide transcription profile of field- and laboratory-selected dichlorodiphenyltrichloroethane (DDT)-resistant Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistasis-el papel esencial de las interacciones de genes en la estructura y la evolución de los sistemas genéticos. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., and Siva-Jothy, M. T. (2003). Inmunología ecológica de los invertebrados. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., y Clark, A. G. (2007). Evolución dinámica del sistema inmunitario innato en Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., and Wertheim, B. (2014). Evolución de una respuesta inmune celular en Drosophila: un análisis comparativo fenotípico y genómico. Genome Biol. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Las redes moleculares como sensores y conductores de enfermedades humanas comunes. Nature 461, 218-223. doi: 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., and Clark, A. G. (2007). Contrasting infection strategies in generalist and specialist wasp parasitoids of Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variación en la defensa inmune como una cuestión de ecología evolutiva. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Ecología evolutiva de las defensas inmunitarias de los insectos. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., y Maniatis, T. (2001). NF-κB vías de señalización en la inmunidad innata de mamíferos e insectos. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., and Clerici, M. (2015). Perspectivas evolutivas de las interacciones huésped-patógeno a partir de datos de secuencias de mamíferos. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Adaptation genomics: the next generation. Trends Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). La secuencia del genoma del bacalao del Atlántico revela un sistema inmunológico único. Nature 477, 207-210. doi: 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Cien años de pleiotropía: una retrospectiva. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., and Wheat, C. W. (2010). Integrando enfoques evolutivos y funcionales para inferir la adaptación en loci específicos. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). La diversidad molecular de la convergencia adaptativa. Science 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., y Tarone, A. M. (2011). La resecuenciación poblacional de poblaciones evolucionadas experimentalmente revela la base genética de la variación del tamaño corporal en Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., y Altenberg, L. (1996). Adaptaciones complejas y la evolución de la evolucionabilidad. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., y Godfray, H. C. (2011). Genómica funcional de la evolución del aumento de la resistencia al parasitismo en Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Genome-wide gene expression in response to parasitoid attack in Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., and Aderem, A. (2014). Análisis a nivel de sistemas de la inmunidad innata. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar