Frontiers in Genetics
Indledning
Mange egenskaber reguleres og koordineres af flere gener og miljøforhold. I naturen udvikler sådanne egenskaber sig løbende under lokalt selektionspres og neutrale processer, hvilket fører til en rig mangfoldighed af fænotypiske varianter og miljømæssige copingstrategier. Allerede Dobzhansky (1964) beskrev den rige mangfoldighed i den levende verden som et resultat af genetisk mangfoldighed, miljømæssig heterogenitet og tilpasninger, der udvikler sig i samspillet mellem molekylær og organismisk biologi. De molekylære mekanismer i evolutionen har været særligt vanskelige at karakterisere. For det første er vi nødt til at forbinde genotypen med fænotypen. Denne forbindelse består af omfattende og indviklede geninteraktionsnetværk (Ayroles et al., 2009; Lehner, 2013). For det andet er vi nødt til at identificere de molekylære ændringer, der er ansvarlige for fænotypiske tilpasninger. Dette afhænger af at belyse den genetiske variation og de genetiske ændringer, der kan forekomme overalt i geninteraktionsnetværkene (f.eks. Edwards et al., 2009).
Den genetiske variations manifestation i gennetværk er meget kompleks. Ændringer i enkelte gener kan påvirke aktiviteten og endda topologien i hele det genetiske netværk (f.eks. Knight et al., 2006). Gener og geninteraktionsnetværk er ofte pleiotropiske og regulerer forskellige egenskaber og processer, hvilket indebærer, at ændringer i et enkelt gen kan manifestere sig i flere egenskaber (Stearns, 2010). Omvendt kan alleliske variationer i mange gener bidrage til variationen i et bestemt fænotypisk træk (Manolio et al., 2009). Desuden er epistase gennemgående, hvilket indebærer, at alleliske variationer på flere loci kan påvirke hinandens fænotypiske virkninger (Phillips, 2008; Mackay og Moore, 2014; Moore og Williams, 2015). Disse overvejelser om det genetiske grundlag for evolution er ikke nye og er blevet undersøgt i flere årtier (f.eks. Wagner og Altenberg, 1996). Forward- og reverse genetik har haft stor succes med at belyse funktionerne af enkelte gener eller mutationer for en bestemt egenskab (Nagy et al., 2003). Disse teknikker er imidlertid begrænsende, når man skal studere kompleksiteten af molekylære interaktionsnetværk, der ligger til grund for en fænotype, eller de molekylære mekanismer for evolutionen af komplekse egenskaber.
Udviklingen inden for genomforskningsteknologi har været et stort løft for vores evne til at studere genetiske kompleksiteter af fænotypiske egenskaber og deres evolution (Stapley et al., 2010). Ved at kombinere disse teknikker med de klassiske genetiske tilgange kan vi vurdere funktionaliteten af genetisk variation for fænotypiske egenskaber (Storz og Wheat, 2010). De første genomiske undersøgelser af flere modelorganismer understregede, at evolutionære tilpasninger, selv for specifikke miljøforhold, generelt styrer mange gener eller loci, samt den dynamiske regulering af genekspressionsmønstre (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). I det følgende årti brugte mange undersøgelser genomforskning til at identificere gener og proteiner, der bidrog til bestemte egenskaber og økologiske interaktioner. I begyndelsen var omkostningerne og den tid, der var nødvendig for at sekventere et enkelt genom, stadig meget restriktive. Next Generation Sequencing har imidlertid gjort det muligt at sekventere genomerne for mange flere arter og mange flere individer pr. art. Dette er en formidabel ressource til at studere evolutionen, da det for første gang gør det muligt for os at kortlægge ændringerne på tværs af hele genomet i løbet af evolutionen.
Genomteknologien har et enormt potentiale til at forbedre vores indsigt i evolutionære processer. Sammenlignende metoder er blevet anvendt til at kortlægge ændringerne i genomsekvenser eller geninteraktionsnetværk på lange evolutionære tidsskalaer (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Eksperimentel udvælgelse eller eksperimentel evolution tilgang, efterfulgt af enten transkriptomik eller genomsekventering, er blevet anvendt til at kortlægge evolutionære ændringer på meget kortere tidsskalaer (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Disse undersøgelser viste for eksempel, hvordan genduplikationer, mutationer og stærk sekvensdivergens i en lille delmængde af gener kan have en dybtgående indvirkning på den transkriptionelle aktivitet af store geninteraktionsnetværk og flere fænotypiske træk.
I dette perspektiv præsenterer jeg de seneste resultater og udviklinger om det genomiske grundlag for evolution, idet jeg bruger udviklende immunitet som et casestudie. Immunitet er en egenskab, der udvikler sig hurtigt, hvilket gør den velegnet til at studere det genomiske grundlag for evolutionære processer (Obbard et al., 2009; Sironi et al., 2015). Desuden er molekylære netværk i immunitet blevet ret godt karakteriseret på grund af deres betydning for menneskers sundhed (Schadt, 2009; Lazzaro og Schneider, 2014; Zak et al., 2014). Først vil jeg meget kort opsummere vores nuværende forståelse af de molekylære netværk af medfødte immunresponser og de udvælgelsesprocesser, der virker på immunresponser. Derefter beskriver jeg de genomiske ændringer, der er forbundet med gevinst, tab og modulering af bestemte aspekter af immunresponser i Drosophila. Endelig foreslår jeg fremtidige retninger for at studere den genetiske arkitektur af komplekse egenskaber og evolutionære processer.
Immunresponser
Immunsystemet består af en kombination af fysiologiske processer, der handler i fællesskab i forsvaret mod patogener og parasitter. Medfødt immunitet er et gammelt træk, der findes i alle flercellede organismer, mens hvirveldyr også besidder erhvervet immunitet. Begge immunsystemer kombinerer cellulære og humorale komponenter: den cellulære komponent omfatter specialiserede celler, der yder en beskyttende funktion. Dette omfatter bl.a. klasser af blodceller til fagocytose af mikrober, indkapsling af større fremmedlegemer eller genkendelse af antigener (i den erhvervede immunitet) og tarmforingen med epitelceller, der danner en fysisk barriere og kan udskille forsvarsforbindelser. Den humorale komponent består af frigivelse af ekstracellulære faktorer, der bekæmper de invaderende patogener, ofte fra specialiserede væv eller cellepopulationer. Dette omfatter frigivelse af antimikrobielle peptider (AMP’er) fra leveren (eller fedtlegemet hos hvirvelløse dyr) og tarmepitelceller, reaktive oxygenarter i fagocytiske og epitelceller og antistoffer fra hvide blodlegemer (i erhvervet immunitet; Lemaitre og Hoffmann, 2007; Buchmann, 2014).
Komplekse netværk af molekylære interaktioner koordinerer immunreaktionerne (Figur 1A). De samme veje er centrale for immunreaktioner fra hvirvelløse dyr til hvirveldyr, hvilket indebærer en stærk bevarelse af kerneelementerne i molekylære netværk i immunitet (Silverman og Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). En række receptormolekyler kan genkende patogener eller parasitter, f.eks. baseret på patogen-associerede molekylære mønstre (f.eks. lipopolysaccharider, der er specifikke for bakteriemembraner). Når receptorer er aktiveret, inducerer de specifikke signaltransduktionsveje, såsom Toll-, Imd- og Jak/Stat-vejene (gennemgået i Lemaitre og Hoffmann, 2007; Buchmann, 2014). Disse veje består af proteaser, kinaser, cytokiner og andre proteiner, som i sidste ende aktiverer transkriptionsfaktorer og co-faktorer. Induktion af disse transkriptionsfaktorer resulterer i produktion af humorale effektormolekyler (f.eks. AMP’er), og det kan inducere proliferation og differentiering af celler, der er involveret i immunitet. Produktionen af forskellige klasser af blodceller er et fremtrædende aspekt af den cellulære komponent af immunresponset, både i den medfødte og erhvervede immunitet. I disse blodceller reguleres også signaltransduktionskaskader for at fremkalde celleegenskaber og proteiner, som bevirker fjernelse af parasitten. For at regulere immunreaktionens styrke, specificitet, timing og varighed moduleres de molekylære netværk af cytokiner, proteaser og kryds og tværs med andre signalveje (Liew et al., 2005; Aggarwal og Silverman, 2008). Dette omfatter også diverse post-transkriptionelle reguleringsnetværk (Ivanov og Anderson, 2013; Carpenter et al., 2014).
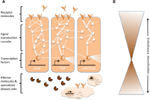
Figur 1. Skematisk repræsentation af de genetiske netværk i immunitet. (A) Flere indbyrdes forbundne netværk koordinerer reaktionerne på en immunudfordring. Disse netværk består af proteiner (repræsenteret ved cirkler), der interagerer med hinanden i en signaltransduktionskaskade for at regulere ekspressionen af transkriptionsfaktorer (repræsenteret ved sekskanter). Aktivering af de centrale signaltransduktionsveje (f.eks. IMD, Toll eller Jak/Stat, angivet ved tykke linjer mellem proteinerne) resulterer i produktion af effektormolekyler som f.eks. antimikrobielle peptider (repræsenteret ved tærteformede symboler) og spredning og differentiering af specialiserede (blod)celler (skyformede figurer). Ekstracellulære og membranbundne receptormolekyler (måneformede figurer) fremkalder disse veje. Aktiviteten kan yderligere moduleres af mange andre proteiner, der interagerer med banerne og krydstaler med andre baner og genetiske netværk (angivet ved de tynde linjer mellem proteinerne). (B) De centrale komponenter i de genetiske netværk i immunitet, f.eks. transkriptionsfaktorerne og de proteiner, der er i direkte kontakt med disse transkriptionsfaktorer, er ofte stærkt bevaret på tværs af stamceller. Evolutionær diversificering findes i højere grad i netværkenes periferi.
Drivkræfterne bag de evolutionære ændringer i immunforsvaret er de kombinerede virkninger af de høje fitnessomkostninger ved infektion for værterne, omkostningerne ved immunitet, den rige diversitet af patogener og parasitter, der truer værterne, og de dynamiske co-evolutionære våbenkapløb mellem værter og patogener (Schmid-Hempel, 2003). Ethvert af disse aspekter eller alle disse aspekter kan virke i værtens lokale miljø og føre til et stærkt selektionspres. Selektionstypen varierer imidlertid fra retningsbestemt over rensende til balancerende, afhængigt af de omkostninger og fordele, som værtspopulationen oplever i sit lokale miljø. F.eks. kan et meget virulent patogen, der invaderer et lokalsamfund, forårsage en selektiv fejning eller retningsbestemt selektion for bestemte resistensalleler, da kun de værter, der har disse alleler, kan bidrage til de næste generationer. Alternativt kan et mangfoldigt eller samudviklende patogensamfund være årsag til frekvensafhængig eller balancerende selektion, der fremmer opretholdelsen af genetisk variation. Udviklingen af immunresponserne afspejler både disse co-evolutionære dynamikker med parasitten og de fysiologiske og økologiske omkostninger ved immunsystemet (Kraaijeveld et al, 2002; Rolff og Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro og Little, 2009).
Genomisk grundlag for evolutionær forandring i Drosophila immunitet
Mens det centrale maskineri af immunresponser er stærkt bevaret, kan flere komponenter af de udvidede molekylære netværk udvikle sig hurtigt eller diversificere sig (Figur 1B). I Drosophila er hurtig evolutionær ændring blevet rapporteret for receptorer og effektorer af immunrespons (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Disse molekyler opererer i grænsefladen mellem værten og patogenet og er derfor afgørende for værtens genkendelse af en invaderende organisme og for at formidle immunresponsets målretning og antagonistiske virkninger på patogenet. Samtidig udvælges parasitten til at forblive uopdaget for at undgå eller afbøde de antagonistiske virkninger af immunresponset. Der forventes derfor en rød dronning-dynamik for disse molekyler i grænsefladen og de molekyler, der modulerer immunforsvaret. Hver part forsøger at få overtaget i det antagonistiske våbenkapløb, hvilket gensidigt medfører ændringer i parternes genetiske netværk. Diversifikationen i receptor-, modulator- og effektormolekyler opnås for det meste ved gen-duplikeringer og hurtige sekvensændringer (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Vi har studeret Drosophilas immunrespons mod parasitoid hvepse som et modelsystem for at forstå det genomiske grundlag for evolutionære processer. Drosophila-larver er værter for en række forskellige parasitoidarter, der lægger æg i disse larver (Fleury et al., 2009). Når parasitoidægget klækkes (∼2-4 dage efter parasitoidangrebet, afhængigt af parasitoidart og temperatur), begynder parasitoidlarven at æde værten og dræber den. Nogle Drosophila-arter har en forsvarsmekanisme mod parasitoider gennem et medfødt immunforsvar, kaldet melanotisk indkapsling. Dette immunrespons består af cellulære og humorale komponenter, der i fællesskab virker for at indkapsle og dræbe parasitoidens æg. Parasitoideangreb udløser immunsignaltransduktionsveje, der inducerer (i) proliferation og differentiering af to klasser af hæmocytter (dvs. insektblodceller), der klæber til parasitoidægget og til hinanden, og (ii) aflejring af melanin på parasitoidægget og den cellulære kapsel omkring parasitoidægget (Lemaitre og Hoffmann, 2007). Værten skal fuldføre den fulde indkapsling og melanisering, før parasitoidægget klækkes, for at overleve parasitoidangrebet.
For genomik-æraen var der blevet identificeret flere gener, der var involveret i immunresponset mod parasitoid hvepse. Toll- og Jak/Stat-vejene var blevet identificeret som centrale komponenter i hæmocytproliferation og -differentiering, og prophenoloxidasevejen for melanisering (gennemgået i Brennan og Anderson, 2004). Der blev derefter gennemført to mikroarray-undersøgelser for at identificere yderligere gener, der potentielt er involveret i den melanotiske indkapsling efter parasitoidangreb. Denne fremgangsmåde fremhævede mange yderligere gener, som ikke tidligere var blevet associeret med immunresponset mod parasitoid, og afslørede deres virkningstidspunkt (Wertheim et al., 2005; Schlenke et al., 2007). Undersøgelserne afslørede flere koordinerede og funktionelt sammenhængende klynger af gener, som midlertidigt blev op- eller nedreguleret under en del af immunresponset (Wertheim et al., 2005). Interessant nok blev det vist, at virulensmekanismerne hos to parasitoidarter adskilte sig fra hinanden i forhold til, hvordan de forstyrrede det genetiske netværk af værtens respons: den ene art eliminerede den indledende aktivering af hele netværket, mens en anden art var rettet mod det sidste trin i kaskaden (Schlenke et al., 2007).
Arter af Drosophila adskiller sig i høj grad i immunitet over for parasitoider. Nogle arter er helt modtagelige over for parasitoider, og dette blev rapporteret som en immundefekt (Eslin og Doury, 2006). En nærmere undersøgelse viste imidlertid, at parasitoidresistens ikke er fælles for alle Drosophila-arter, men er begrænset til nogle få klasser. Vi viste, at i en af disse klader, melanogaster-undergruppen, var den evolutionære gevinst af parasitoidresistens forbundet med gevinsten af en ny type blodcelle, lamellocytterne, som også er begrænset til den samme klade (Salazar-Jaramillo et al., 2014). Nogle Drosophila-arter uden for melanogaster-undergruppen kan også indkapsle parasitoidæg, men de synes at have udviklet forskellige typer af blodceller til indkapslingsreaktionen (Havard et al., 2012; Márkus et al., 2015). Immunresponset mod parasitoider har udviklet sig uafhængigt af hinanden i forskellige insekttaxaer, ofte med lidt forskellige mekanismer og typer af blodceller (Lavine og Strand, 2002). Evolutionen af det medfødte immunsystem omfatter således tilføjelse af nye komponenter eller “moduler”. Dette rejser spørgsmålet om, hvordan genomet ændrer sig under tilegnelsen af et nyt modul.
Sammenlignende genomik afslørede, at på trods af tilegnelsen af en ny type blodcelle i melanogaster-undergruppen, er de gener, der er kendt for lamellocytdifferentiering, stort set bevaret på tværs af hele fylogenien. Også arter, der ikke producerer lamellocytter som reaktion på parasitoidangreb, besidder disse gener, der er nødvendige for lamellocytdifferentiering. Desuden viser disse gener kun lidt divergens eller signaturer af selektion, mens det ville være forventeligt for gener, der har fået en ny funktion. Dette tyder på, at de eksisterende signaltransduktionsveje for hæmocytdifferentiering bliver moduleret af det omgivende geninteraktionsnetværk for at producere en ny type blodcelle i melanogaster-undergruppen. Denne sammentænkning af den eksisterende kernehæmocytproliferationsvej for hæmocytter opnås sandsynligvis ved at tilføje andre eller nye komponenter til geninteraktionsnetværket (Salazar-Jaramillo et al., 2014). Vi identificerede flere nye gener, der opstod omkring tidspunktet for erhvervelse af lamellocytter og er differentielt udtrykt under immunresponset mod parasitoider, herunder receptormolekyler og proteaser af serintypen (Salazar-Jaramillo et al., 2014). Vi stiller den hypotese, at især serintype endopeptidaser kan spille en afgørende rolle i denne udvidelse af geninteraktionsnetværket. Et betydeligt antal af disse molekyler opstod på tidspunktet for erhvervelse af lamellocytter, de udtrykkes på det rigtige tidspunkt i immunresponset, og de viser stærke signaturer af positiv udvælgelse (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
Også inden for en enkelt art, D. melanogaster, viser immunresponser stor genetisk variation. Feltpopulationer indsamlet fra hele Europa viser betydelige forskelle i evnen til at indkapsle parasitoidæg med succes (Kraaijeveld og van Alphen, 1995; Kraaijeveld og Godfray, 1999; Gerritsma et al., 2013). Tilsyneladende er omkostningerne og fordelene ved et stærkt immunforsvar geografisk forskellige, hvilket fører til modulering og differentiering af co-tilpassede genetiske netværk. Dette blev også afspejlet i den hæmocytiske reaktion efter parasitoidangreb. Feltlinjerne varierede betydeligt i det absolutte og relative antal af de forskellige hæmocytter, som de producerede som reaktion på parasitoidangreb, selv blandt de linjer, der havde stor succes med indkapsling (Gerritsma et al., 2013). Dette understreger igen, at den genetiske baggrund i en population og det kombinerede lokale selektionspres fører til alternative evolutionære reaktioner. Sammenligning af genomerne af resistente og modtagelige individer fra flere populationer kan afsløre den adaptive variation i den genetiske arkitektur af denne egenskab.
For at kortlægge ændringerne i genomet under evolutionen af øget resistens, gennemførte vi eksperimentel evolution for parasitoidresistens. I laboratoriet udsatte vi en stor udvokset population for parasitoider. Kun de larver, der formåede at overleve parasitoidangreb, fik lov til at bidrage til den næste generation. Med denne fremgangsmåde øgede vi resistensniveauet fra 20 til ∼50 % af de larver, der overlevede parasitoidangreb, efter kun fem generationers udvælgelse. Da vi målte ændringer i genekspressionen i de udvalgte populationer sammenlignet med genekspressionen i kontrollinjerne, selv før parasitoidangreb, fandt vi flere hundrede gener, som var let differentielt reguleret (Wertheim et al., 2011). Ændringerne involverede hovedsageligt gener, der ikke blev differentielt udtrykt under immunresponset, hvilket indikerer, at de evolutionære ændringer ikke præ-aktiverede immunresponset i forventning om parasitoidangreb, men at de modulerede den kanoniske udviklingsvej, hvilket (også) førte til en forøgelse af dens forsvarsevner. Vi gentog dette eksperiment og sekventerede derefter genomerne af de udvalgte og kontrolpopulationerne. I genomerne af de linjer, der udviklede øget modstandsdygtighed, fandt vi signaturer af udvælgelse på flere snævert definerede regioner af genomet (Jalvingh et al., 2014). Nogle af disse regioner overlappede også med de regioner, der viste ændret udtryk efter udvælgelse for øget resistens (Wertheim et al., 2011; Jalvingh et al., 2014). Således kan et hurtigt og stærkt selektivt sweep på en kompleks egenskab som immunitet stadig påvirke flere, men meget lokaliserede, genomiske regioner.
Fremtidige udfordringer
Hvordan skal vi forene de langsigtede evolutionære ændringer, såsom erhvervelse af nye gener i geninteraktionsnetværk, og de kortsigtede evolutionære ændringer, såsom sekvensvarianter, der kan fejes gennem en population? Nøglen hertil er at (i) rekonstruere det geninteraktionsnetværk, der ligger til grund for komplekse egenskaber, og (ii) karakterisere den rolle, som genetisk variation spiller i disse netværk. Genetiske netværk kan udvides med nye gener gennem f.eks. duplikationer, blive forbundet med andre netværk eller moduler, og små sekvensvariationer kan modulere netværkenes aktivitet og topologi. Hvis vi kan dekomponere de genetiske netværk og vurdere den rolle, som genetiske varianter spiller i en netværkskontekst, vil dette i sidste ende gøre det muligt for os at afgøre, hvordan genetisk variation omsættes til fænotypisk variation. Dette vil også forbedre vores forståelse af det molekylære grundlag for komplekse menneskelige sygdomme og udviklingen af medfødt og erhvervet immunitet (Cooper og Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay og Moore, 2014; Sironi et al., 2015).
Systembiologiske tilgange vil være uvurderlige til at afdække de komplekse geninteraktionsnetværk. Der udvikles matematiske modeller til at beskrive de molekylære mekanismer, der ligger til grund for en egenskab, og til at forudsige dynamikken i grupper af interagerende komponenter i netværket. Modellerne er baseret på molekylærgenetiske og genomiske data. På nuværende tidspunkt anvendes systembiologi mest på specifikke træk i encellede organismer, og dette anses for at være grænsen for, hvad der kan opnås (Papp et al., 2011). Det er dog sandsynligt, at denne model ikke er repræsentativ for evolutionen i kønsligt reproducerende flercellede organismer. I stedet for at vente på, at de forenklede modeller nøjagtigt afspejler små undernetværk, skal vi udvikle og forfine vores metoder til at udnytte og kvantificere de emergente egenskaber fra den store mængde genomdata. Vi kan udlede geninteraktionsnetværk fra protein-protein- eller transkriptkorrelation eller koekspressionsmatricer (Shannon et al., 2003; Langfelder og Horvath, 2008) og sammensmelte disse med analyser af naturlige varianter (Nuzhdin et al., 2012). Vi bør videreudvikle disse metoder og alternative tilgange for fuldt ud at udnytte vores målinger på genomdata og for at konvertere disse kvantitative målinger til netværksanalyser. Mens genomdata i sig selv ikke vil give det fulde svar på, hvad der bestemmer livets adaptive kapacitet, giver de os mulighed for at kvantificere og observere, hvad der sker på molekylært niveau i løbet af evolutionen. Når vi kombinerer og integrerer dette med miljøets heterogenitet som drivkraft for tilpasninger, vil vi måske kunne afsløre de komplekse molekylære mekanismer for tilpasning og evolution.
Interessekonflikterklæring
Forfatteren erklærer, at forskningen blev udført i fravær af kommercielle eller finansielle relationer, der kunne opfattes som en potentiel interessekonflikt.
Akkreditering
Jeg takker Kirsten Jalvingh, Laura Salazar-Jaramillo og Sylvia Gerritsma for værdifulde diskussioner. BW blev støttet af midler fra den nederlandske organisation for videnskabelig forskning (NWO; bevilling 864.08.008).
Aggarwal, K., og Silverman, N. (2008). Positiv og negativ regulering af Drosophila immunsvaret. BMB Rep. 41, 267-277. doi: 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M. M., et al. (2009). Systemgenetik af komplekse egenskaber i Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., og Anderson, K. V. (2004). Drosophila: genetik af medfødt immungenkendelse og -respons. Annu. Rev. Immunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evolution af medfødt immunitet: ledetråde fra hvirvelløse dyr via fisk til pattedyr. Front. Immunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., og Fitzgerald, K. A. (2014). Post-transkriptionel regulering af genekspression i den medfødte immunitet. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., og Alder, M. N. (2006). Udviklingen af adaptive immunsystemer. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biologi, molekylær og organismisk. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Evolution af gener og genomer i Drosophila-fylogenien. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., og Mackay, T. F. C. (2009). Et transkriptionelt netværk forbundet med naturlig variation i Drosophila aggressiv adfærd. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., og Doury, G. (2006). Fluen Drosophila subobscura: et naturligt tilfælde af mangel på medfødt immunitet. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., og Banerjee, U. (2003). Tykkere end blod: Konserverede mekanismer i Drosophila og hvirveldyrs hæmatopoiesis. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., H. L., Sniegowski, P. D., og Eisen, M. B. (2004). Populationsgenetisk variation i genekspression er forbundet med fænotypisk variation i Saccharomyces cerevisiae. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., og Allemand, R. (2009). Økologi og livshistorieudvikling hos frugivoriske Drosophila-parasitoider. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Genomiske ekspressionsprogrammer i gærcellers reaktion på miljøændringer. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., og Wertheim, B. (2013). Naturlig variation i differentierede hemocytter er relateret til parasitoidresistens hos Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., og Eslin, P. (2012). Strukturel og funktionel karakterisering af pseudopodocyte, en shaggy immuncelle produceret af to Drosophila-arter af obscura-gruppen. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). Eksperimentel evolution, genetisk analyse og genome re-sequencing afslører den mutation, der giver artemisininresistens i en isogen slægt af malariaparasitter. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., og Anderson, P. (2013). Post-transkriptionelle reguleringsnetværk i immunitet. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., og Wertheim, B. (2014). Genomiske ændringer under hurtig evolution: selektion for parasitoidresistens. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). Det genomiske grundlag for adaptiv evolution hos trefinnede stimefisk. Nature 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Afsløring af adaptiv evolution: hvordan en enkelt punktmutation påvirker proteinkoreguleringsnetværket. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., og Godfray, H. C. J. (1999). Geografiske mønstre i udviklingen af resistens og virulens hos Drosophila og dens parasitoider. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., og Godfray, H. C. J. (2002). Omkostninger ved resistens i interaktioner mellem insekt-parasit og insekt-parasitoid. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., og van Alphen, J. J. M. M. (1995). Geografisk variation i indkapslingsevne hos Drosophila melanogaster-larver og beviser for parasitoidspecifikke komponenter. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., og Horvath, S. (2008). WGCNA: en R-pakke til vægtet korrelationsnetværksanalyse. BMC Bioinformatics 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., og Strand, M. R. (2002). Insekthæmocytter og deres rolle i immunitet. Insect Biochem. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., og Little, T. J. (2009). Immunitet i en variabel verden. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., og Schneider, D. S. (2014). Immunitetens genetik. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Genotype til fænotype: erfaringer fra modelorganismer for humangenetik. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., og Hoffmann, J. (2007). Værtsforsvaret hos Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., og O’Neill, L. A. J. (2005). Negativ regulering af Toll-like receptor-medierede immunresponser. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). Adaptiv evolution af flere egenskaber gennem flere mutationer på et enkelt gen. Science 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., og Moore, J. H. (2014). Hvorfor epistase er vigtig for håndtering af kompleks menneskelig sygdomsgenetik. Genome Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). At finde den manglende arvelighed af komplekse sygdomme. Nature 461, 747-753. doi: 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Multinucleated giant hemocytes er effektorceller i cellemedierede immunresponser hos Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., og Williams, S. M. (eds). (2015). Epistasis. New York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., og Plasterk, R. (2003). Skræddersyning af genomet: kraften i genetiske metoder. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., og Stubbs, L. (2009). Forskelle i menneskers og chimpansers genekspressionsmønstre definerer et udviklende netværk af transkriptionsfaktorer i hjernen. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., og McIntyre, L. M. (2012). Genotype-fænotype-kortlægning i en post-GWAS-verden. Trends Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J. J., Kim, K.-W., og Jiggins, F. M. (2009). Kvantificering af adaptiv evolution i Drosophila-immunforsvaret. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., og Pál, C. (2011). Systembiologiske tilgange til forudsigelse af genomisk evolution. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., og Pittendrigh, B. R. (2004). Genome-wide transkriptionsprofil af felt- og laboratorie-selekteret dichlordiphenyltrichloroethan (DDT)-resistent Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.040058010101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistase – den afgørende rolle af geninteraktioner i strukturen og evolutionen af genetiske systemer. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., and Siva-Jothy, M. T. (2003). Hvirvelløse dyrs økologiske immunologi. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., og Clark, A. G. (2007). Dynamisk udvikling af det medfødte immunsystem i Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., og Wertheim, B. (2014). Evolution af et cellulært immunrespons i Drosophila: en fænotypisk og genomisk komparativ analyse. Genome Biol. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Molekylære netværk som sensorer og drivkræfter for almindelige menneskelige sygdomme. Nature 461, 218-223. doi: 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., og Clark, A. G. (2007). Kontrasterende infektionsstrategier hos generalist- og specialiserede hvepseparasitoider af Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variation i immunforsvaret som et spørgsmål om evolutionær økologi. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Evolutionær økologi af insekters immunforsvar. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: et softwaremiljø til integrerede modeller af biomolekylære interaktionsnetværk. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., og Maniatis, T. (2001). NF-κB-signalveje i pattedyrs og insekters medfødte immunitet. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., og Clerici, M. (2015). Evolutionær indsigt i vært-patogene interaktioner fra sekvensdata fra pattedyr. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Tilpasningsgenomik: den næste generation. Trends Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). Genomsekvensen af atlanterhavstorsk afslører et unikt immunsystem. Nature 477, 207-210. doi: 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Et hundrede år med pleiotropi: et retrospektivt. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., og Wheat, C. W. (2010). Integrering af evolutionære og funktionelle tilgange til at udlede tilpasning på specifikke loci. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). Den molekylære mangfoldighed af adaptiv konvergens. Science 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., og Tarone, A. M. (2011). Populationsbaseret resekventering af eksperimentelt udviklede populationer afslører det genetiske grundlag for variation i kropsstørrelse hos Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., og Altenberg, L. (1996). Komplekse tilpasninger og udviklingen af udviklingsmuligheder. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., og Godfray, H. C. (2011). Funktionel genomik af evolutionen af øget modstandsdygtighed over for parasitisme hos Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Genome-wide genekspression som reaktion på parasitoidangreb hos Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., og Aderem, A. (2014). Analyse på systemniveau af den medfødte immunitet. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar