Frontiers in Genetics
Introduction
Viele Merkmale werden von mehreren Genen und Umweltbedingungen reguliert und koordiniert. In der Natur entwickeln sich solche Merkmale unter lokalem Selektionsdruck und neutralen Prozessen kontinuierlich weiter, was zu einer großen Vielfalt an phänotypischen Varianten und Umweltbewältigungsstrategien führt. Bereits Dobzhansky (1964) beschrieb die reiche Vielfalt in der Welt der Lebewesen als Ergebnis der genetischen Vielfalt, der Heterogenität der Umwelt und der Anpassungen, die sich im Zusammenspiel von Molekular- und Organismusbiologie entwickeln. Die molekularen Mechanismen der Evolution sind besonders schwer zu charakterisieren. Erstens müssen wir eine Verbindung zwischen dem Genotyp und dem Phänotyp herstellen. Diese Verbindung besteht aus umfangreichen und komplizierten Geninteraktionsnetzwerken (Ayroles et al., 2009; Lehner, 2013). Zweitens müssen wir die molekularen Veränderungen identifizieren, die für phänotypische Anpassungen verantwortlich sind. Dies hängt von der Aufklärung der genetischen Variation und der genetischen Veränderungen ab, die überall in den Geninteraktionsnetzwerken auftreten können (z. B. Edwards et al., 2009).
Die Manifestation der genetischen Variation in Gennetzwerken ist sehr komplex. Veränderungen in einzelnen Genen können die Aktivität und sogar die Topologie des gesamten genetischen Netzwerks beeinflussen (z. B. Knight et al., 2006). Gene und Geninteraktionsnetzwerke sind häufig pleiotrop und regulieren verschiedene Merkmale und Prozesse, was bedeutet, dass sich Veränderungen in einem einzelnen Gen in mehreren Merkmalen manifestieren können (Stearns, 2010). Umgekehrt können allele Variationen in vielen Genen zur Variation in einem bestimmten phänotypischen Merkmal beitragen (Manolio et al., 2009). Darüber hinaus ist die Epistase weit verbreitet, was bedeutet, dass allelische Variationen an mehreren Loci die phänotypischen Auswirkungen der jeweils anderen beeinflussen können (Phillips, 2008; Mackay und Moore, 2014; Moore und Williams, 2015). Diese Überlegungen zu den genetischen Grundlagen der Evolution sind nicht neu und werden seit mehreren Jahrzehnten untersucht (z. B. Wagner und Altenberg, 1996). Vorwärts- und Rückwärtsgenetik haben sich bei der Aufklärung der Funktionen einzelner Gene oder Mutationen für ein bestimmtes Merkmal als äußerst erfolgreich erwiesen (Nagy et al., 2003). Diese Techniken sind jedoch bei der Untersuchung der Komplexität molekularer Interaktionsnetzwerke, die einem Phänotyp zugrunde liegen, oder der molekularen Mechanismen der Evolution komplexer Merkmale begrenzt.
Die Entwicklungen in der Genomik-Technologie haben unsere Fähigkeit, die genetische Komplexität phänotypischer Merkmale und deren Evolution zu untersuchen, erheblich verbessert (Stapley et al., 2010). Die Kombination dieser Techniken mit den klassischen genetischen Ansätzen ermöglicht es uns, die Funktionalität der genetischen Variation für phänotypische Merkmale zu bewerten (Storz und Wheat, 2010). Die ersten genomischen Studien an verschiedenen Modellorganismen betonten, dass evolutionäre Anpassungen, selbst an spezifische Umweltbedingungen, im Allgemeinen viele Gene oder Loci sowie die dynamische Regulierung von Genexpressionsmustern steuern (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). In den folgenden zehn Jahren wurde die Genomik in vielen Studien zur Identifizierung von Genen und Proteinen eingesetzt, die zu bestimmten Merkmalen und ökologischen Interaktionen beitragen. Anfänglich waren die Kosten und der Zeitaufwand für die Sequenzierung eines einzelnen Genoms noch sehr restriktiv. Mit dem Next Generation Sequencing ist es jedoch möglich geworden, die Genome von viel mehr Arten und viel mehr Individuen pro Art zu sequenzieren. Dies ist eine hervorragende Ressource für die Erforschung der Evolution, da es uns zum ersten Mal ermöglicht, die Veränderungen im gesamten Genom während der Evolution zu kartieren.
Die Genomik-Technologie hat ein enormes Potenzial, unsere Erkenntnisse über evolutionäre Prozesse zu verbessern. Vergleichende Ansätze wurden angewandt, um die Veränderungen in den Genomsequenzen oder Geninteraktionsnetzwerken auf langen evolutionären Zeitskalen zu kartieren (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Experimentelle Selektion oder experimentelle Evolutionsansätze, gefolgt von Transkriptomik oder Genomsequenzierung, wurden verwendet, um evolutionäre Veränderungen auf viel kürzeren Zeitskalen zu kartieren (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Diese Studien haben beispielsweise gezeigt, wie Genduplikationen, Mutationen und starke Sequenzdivergenzen in einer kleinen Untergruppe von Genen einen tiefgreifenden Einfluss auf die transkriptionelle Aktivität großer Geninteraktionsnetzwerke und mehrere phänotypische Merkmale haben können.
In dieser Perspektive stelle ich die jüngsten Erkenntnisse und Entwicklungen auf dem Gebiet der genomischen Grundlagen der Evolution vor, wobei ich die sich entwickelnde Immunität als Fallstudie verwende. Immunität ist ein Merkmal, das sich schnell entwickelt und sich daher gut für die Untersuchung der genomischen Grundlagen von Evolutionsprozessen eignet (Obbard et al., 2009; Sironi et al., 2015). Darüber hinaus sind die molekularen Netzwerke der Immunität aufgrund ihrer Bedeutung für die menschliche Gesundheit recht gut charakterisiert worden (Schadt, 2009; Lazzaro und Schneider, 2014; Zak et al., 2014). Zunächst fasse ich kurz unser derzeitiges Verständnis der molekularen Netzwerke angeborener Immunantworten und der Selektionsprozesse, die auf Immunantworten wirken, zusammen. Dann beschreibe ich die genomischen Veränderungen, die mit dem Gewinn, dem Verlust und der Modulation bestimmter Aspekte der Immunreaktionen in Drosophila verbunden sind. Schließlich schlage ich künftige Richtungen vor, um die genetische Architektur komplexer Merkmale und evolutionärer Prozesse zu untersuchen.
Immunreaktionen
Das Immunsystem besteht aus einer Kombination physiologischer Prozesse, die gemeinsam bei der Abwehr von Krankheitserregern und Parasiten wirken. Die angeborene Immunität ist eine uralte Eigenschaft, die bei allen vielzelligen Organismen zu finden ist, während Wirbeltiere auch eine erworbene Immunität besitzen. Beide Immunsysteme kombinieren zelluläre und humorale Komponenten: Die zelluläre Komponente umfasst spezialisierte Zellen, die eine Schutzfunktion ausüben. Dazu gehören Klassen von Blutzellen für die Phagozytose von Mikroben, die Einkapselung größerer Fremdkörper oder die Erkennung von Antigenen (bei der erworbenen Immunität) sowie die Auskleidung des Darms mit Epithelzellen, die eine physische Barriere bilden und Abwehrstoffe absondern können. Die humorale Komponente besteht in der Freisetzung von extrazellulären Faktoren, die die eindringenden Krankheitserreger bekämpfen, häufig aus spezialisierten Geweben oder Zellpopulationen. Dazu gehören die Freisetzung antimikrobieller Peptide (AMPs) durch die Leber (oder den Fettkörper bei Wirbellosen) und Darmepithelzellen, reaktive Sauerstoffspezies in phagozytären und epithelialen Zellen sowie Antikörper aus weißen Blutkörperchen (bei der erworbenen Immunität; Lemaitre und Hoffmann, 2007; Buchmann, 2014).
Komplexe Netzwerke molekularer Interaktionen koordinieren die Immunantworten (Abbildung 1A). Dieselben Signalwege sind für Immunantworten von Wirbellosen bis zu Wirbeltieren von zentraler Bedeutung, was bedeutet, dass die Kernelemente der molekularen Netzwerke in der Immunität weitgehend erhalten bleiben (Silverman und Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). Eine Vielzahl von Rezeptormolekülen kann Krankheitserreger oder Parasiten erkennen, z. B. anhand von pathogenassoziierten molekularen Mustern (z. B. Lipopolysaccharide, die spezifisch für bakterielle Membranen sind). Sobald die Rezeptoren aktiviert sind, lösen sie spezifische Signaltransduktionswege aus, wie z. B. die Toll-, Imd- und Jak/Stat-Wege (siehe Lemaitre und Hoffmann, 2007; Buchmann, 2014). Diese Signalwege bestehen aus Proteasen, Kinasen, Zytokinen und anderen Proteinen, die schließlich Transkriptionsfaktoren und Kofaktoren aktivieren. Die Induktion dieser Transkriptionsfaktoren führt zur Produktion humoraler Effektormoleküle (z. B. AMPs) und kann die Proliferation und Differenzierung von an der Immunität beteiligten Zellen induzieren. Die Produktion verschiedener Klassen von Blutzellen ist ein wichtiger Aspekt der zellulären Komponente der Immunantwort, sowohl bei der angeborenen als auch bei der erworbenen Immunität. In diesen Blutzellen werden auch Signaltransduktionskaskaden reguliert, um Zelleigenschaften und Proteine zu induzieren, die die Beseitigung des Parasiten bewirken. Um die Stärke, Spezifität, den Zeitpunkt und die Dauer von Immunreaktionen zu regulieren, werden die molekularen Netzwerke durch Zytokine, Proteasen und Überkreuzverbindungen mit anderen Signalwegen moduliert (Liew et al., 2005; Aggarwal und Silverman, 2008). Dazu gehören auch diverse posttranskriptionelle regulatorische Netzwerke (Ivanov und Anderson, 2013; Carpenter et al., 2014).
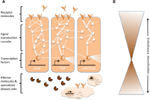
Abbildung 1. Schematische Darstellung der genetischen Netzwerke in der Immunität. (A) Mehrere miteinander verbundene Netzwerke koordinieren die Reaktionen auf eine Immunreaktion. Diese Netzwerke bestehen aus Proteinen (dargestellt durch Kreise), die in einer Signaltransduktionskaskade miteinander interagieren, um die Expression von Transkriptionsfaktoren (dargestellt durch Sechsecke) zu regulieren. Die Aktivierung der wichtigsten Signaltransduktionswege (z. B. IMD, Toll oder Jak/Stat, dargestellt durch dicke Linien zwischen den Proteinen) führt zur Produktion von Effektormolekülen wie antimikrobiellen Peptiden (dargestellt durch kuchenförmige Symbole) und zur Vermehrung und Differenzierung spezialisierter (Blut-)Zellen (wolkenförmige Figuren). Extrazelluläre und membrangebundene Rezeptormoleküle (mondförmige Figuren) induzieren die Signalwege. Die Aktivität kann darüber hinaus durch viele andere Proteine moduliert werden, die mit den Signalwegen interagieren und mit anderen Signalwegen und genetischen Netzwerken in Wechselwirkung stehen (angedeutet durch die dünnen Linien zwischen den Proteinen). (B) Die zentralen Komponenten der genetischen Netzwerke in der Immunität, z. B. die Transkriptionsfaktoren und die Proteine, die in direktem Kontakt mit diesen Transkriptionsfaktoren stehen, sind häufig phylogenieübergreifend stark konserviert. Die evolutionäre Diversifizierung findet eher in den Randbereichen der Netzwerke statt.
Die Triebkräfte für die evolutionären Veränderungen in den Immunantworten sind die kombinierten Auswirkungen der hohen Fitnesskosten der Infektion auf die Wirte, die Kosten der Immunität, die reiche Vielfalt an Pathogenen und Parasiten, die die Wirte bedrohen, und das dynamische ko-evolutionäre Wettrüsten zwischen Wirten und Pathogenen (Schmid-Hempel, 2003). Jeder oder alle diese Aspekte können in der lokalen Umgebung des Wirts wirken und zu einem starken Selektionsdruck führen. Die Art der Selektion variiert jedoch von gerichteter über reinigende bis hin zu ausgleichender Selektion, abhängig von den Kosten und Vorteilen, die die Wirtspopulation in ihrer lokalen Umgebung erfährt. So kann beispielsweise ein hochvirulenter Erreger, der in eine lokale Gemeinschaft eindringt, einen selektiven Sweep oder eine gerichtete Selektion für bestimmte Resistenzallele verursachen, da nur die Wirte mit diesen Allelen zur nächsten Generation beitragen können. Andererseits kann eine vielfältige oder sich gemeinsam entwickelnde Gemeinschaft von Krankheitserregern eine frequenzabhängige oder ausgleichende Selektion bewirken, die die Aufrechterhaltung der genetischen Variation begünstigt. Die Evolution der Immunreaktionen spiegelt sowohl diese koevolutionäre Dynamik mit dem Parasiten als auch die physiologischen und ökologischen Kosten des Immunsystems wider (Kraaijeveld et al., 2002; Rolff und Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro und Little, 2009).
Genomische Grundlagen der evolutionären Veränderungen in der Drosophila-Immunität
Während die zentrale Maschinerie der Immunantworten stark konserviert ist, können sich mehrere Komponenten der erweiterten molekularen Netzwerke schnell entwickeln oder diversifizieren (Abbildung 1B). Bei Drosophila wurde ein schneller evolutionärer Wandel bei den Rezeptoren und Effektoren der Immunreaktion festgestellt (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Diese Moleküle wirken an der Schnittstelle zwischen dem Wirt und dem Erreger und sind daher entscheidend für die Erkennung eines eindringenden Organismus durch den Wirt und für die Vermittlung der zielgerichteten und antagonistischen Wirkung der Immunantwort auf den Erreger. Gleichzeitig steht der Parasit unter der Selektion, unentdeckt zu bleiben, um die antagonistischen Wirkungen der Immunreaktion zu vermeiden oder abzuschwächen. Daher wird für diese Moleküle an der Schnittstelle und für die Moleküle, die die Immunreaktionen modulieren, eine Dynamik der roten Königin erwartet. Jede Partei versucht, in dem antagonistischen Wettrüsten die Oberhand zu gewinnen, was wechselseitig zu Veränderungen in den genetischen Netzwerken der Parteien führt. Die Diversifizierung der Rezeptor-, Modulator- und Effektormoleküle wird meist durch Genduplikationen und schnelle Sequenzänderungen erreicht (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Wir haben die Immunantwort von Drosophila gegen parasitische Wespen als Modellsystem untersucht, um die genomischen Grundlagen evolutionärer Prozesse zu verstehen. Drosophila-Larven sind Wirt für eine Vielzahl von Parasitoidenarten, die ein Ei in diese Larven legen (Fleury et al., 2009). Sobald das Parasitoiden-Ei geschlüpft ist (∼2-4 Tage nach dem Parasitoidenbefall, je nach Parasitoidenart und Temperatur), beginnt die Parasitoiden-Larve mit dem Fressen des Wirts und tötet ihn. Einige Drosophila-Arten verfügen über einen Abwehrmechanismus gegen Parasitoide, der auf einer angeborenen Immunreaktion beruht und als melanotische Verkapselung bezeichnet wird. Diese Immunreaktion besteht aus zellulären und humoralen Komponenten, die zusammenwirken, um das Parasitoiden-Ei abzusondern und zu töten. Der Angriff der Parasitoiden löst Immunsignalwege aus, die (i) die Vermehrung und Differenzierung von zwei Klassen von Hämozyten (d. h. Insektenblutzellen), die sich an das Parasitoiden-Ei und aneinander heften, und (ii) die Ablagerung von Melanin auf dem Parasitoiden-Ei und der Zellkapsel um das Parasitoiden-Ei herum auslösen (Lemaitre und Hoffmann, 2007). Der Wirt muss die vollständige Verkapselung und Melanisierung abschließen, bevor das Parasitoiden-Ei schlüpft, um den Parasitoiden-Befall zu überleben.
Vor der Ära der Genomik wurden mehrere Gene identifiziert, die an der Immunantwort gegen parasitoide Wespen beteiligt sind. Die Toll- und Jak/Stat-Signalwege waren als zentrale Komponenten der Hämozytenproliferation und -differenzierung identifiziert worden, und der Prophenoloxidase-Signalweg für die Melanisierung (Übersicht in Brennan und Anderson, 2004). Anschließend wurden zwei Microarray-Studien durchgeführt, um weitere Gene zu identifizieren, die möglicherweise an der melanotischen Verkapselung nach einem Parasitoidenbefall beteiligt sind. Dieser Ansatz förderte viele zusätzliche Gene zutage, die bisher nicht mit der Immunreaktion gegen Parasitoiden in Verbindung gebracht worden waren, und offenbarte deren Wirkungszeitpunkt (Wertheim et al., 2005; Schlenke et al., 2007). Die Studien ergaben mehrere koordinierte und funktionell kohärente Gruppen von Genen, die während eines Teils der Immunreaktion vorübergehend hoch- oder herunterreguliert wurden (Wertheim et al., 2005). Interessanterweise konnte gezeigt werden, dass sich die Virulenzmechanismen zweier Parasitoidenarten darin unterscheiden, wie sie in das genetische Netzwerk der Wirtsreaktionen eingreifen: eine Art schaltete die anfängliche Aktivierung des gesamten Netzwerks aus, während eine andere Art auf den letzten Schritt der Kaskade abzielte (Schlenke et al., 2007).
Drosophila-Arten unterscheiden sich stark in ihrer Immunität gegen Parasitoide. Einige Arten sind für Parasitoide völlig unempfänglich, was als Immunschwäche bezeichnet wurde (Eslin und Doury, 2006). Bei näherer Betrachtung stellte sich jedoch heraus, dass die Parasitoidenresistenz nicht allen Drosophila-Arten gemeinsam ist, sondern auf einige wenige Gruppen beschränkt ist. Wir haben gezeigt, dass in einer dieser Gruppen, der Melanogaster-Untergruppe, der evolutionäre Zugewinn an Parasitenresistenz mit dem Zugewinn einer neuen Art von Blutzellen, den Lamellozyten, verbunden war, die ebenfalls auf dieselbe Gruppe beschränkt ist (Salazar-Jaramillo et al., 2014). Einige Drosophila-Arten außerhalb der Melanogaster-Untergruppe können ebenfalls Parasitoiden-Eier einkapseln, aber sie scheinen andere Blutzelltypen für die Einkapselungsreaktion entwickelt zu haben (Havard et al., 2012; Márkus et al., 2015). Die Immunantwort gegen Parasitoide hat sich in verschiedenen Insektentaxa unabhängig voneinander entwickelt, oft mit leicht unterschiedlichen Mechanismen und Blutzelltypen (Lavine und Strand, 2002). Die Evolution des angeborenen Immunsystems umfasst also auch das Hinzufügen neuer Komponenten oder „Module“. Dies wirft die Frage auf, wie sich das Genom während des Erwerbs eines neuen Moduls verändert.
Vergleichende Genomik ergab, dass trotz des Zugewinns eines neuen Blutzelltyps in der Melanogaster-Untergruppe die Gene, die für die Lamellocyten-Differenzierung bekannt sind, über die gesamte Phylogenie weitgehend konserviert sind. Auch Arten, die als Reaktion auf Parasitoidenbefall keine Lamellozyten produzieren, besitzen diese Gene, die für die Lamellozytendifferenzierung erforderlich sind. Darüber hinaus weisen diese Gene kaum Divergenz oder Selektionssignaturen auf, was bei Genen, die eine neue Funktion erhalten haben, zu erwarten wäre. Dies deutet darauf hin, dass die bestehenden Signaltransduktionswege für die Hämozytendifferenzierung durch das umgebende Geninteraktionsnetzwerk moduliert werden, um einen neuartigen Blutzelltyp in der Melanogaster-Untergruppe zu erzeugen. Diese Kooptation des bestehenden zentralen Hämozytenproliferationswegs wird wahrscheinlich durch das Hinzufügen anderer oder neuer Komponenten zum Geninteraktionsnetzwerk erreicht (Salazar-Jaramillo et al., 2014). Wir haben mehrere neue Gene identifiziert, die um die Zeit des Lamellocytenerwerbs entstanden sind und während der Immunantwort gegen Parasitoide unterschiedlich exprimiert werden, darunter Rezeptormoleküle und Proteasen vom Serintyp (Salazar-Jaramillo et al., 2014). Wir stellen die Hypothese auf, dass insbesondere die Endopeptidasen vom Serintyp eine entscheidende Rolle bei dieser Erweiterung des Geninteraktionsnetzwerks spielen könnten. Eine beträchtliche Anzahl dieser Moleküle entstand zum Zeitpunkt des Erwerbs von Lamellozyten, sie werden zum richtigen Zeitpunkt in der Immunantwort exprimiert und sie zeigen starke Anzeichen einer positiven Selektion (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
Auch innerhalb einer einzigen Art, D. melanogaster, zeigen Immunantworten eine große genetische Variation. Feldpopulationen, die in ganz Europa gesammelt wurden, zeigen erhebliche Unterschiede in der Fähigkeit, Parasitoiden-Eier erfolgreich einzukapseln (Kraaijeveld und van Alphen, 1995; Kraaijeveld und Godfray, 1999; Gerritsma et al., 2013). Offensichtlich sind die Kosten und Vorteile einer starken Immunabwehr geografisch unterschiedlich, was zu einer Modulation und Differenzierung ko-angepasster genetischer Netzwerke führt. Dies spiegelte sich auch in der hämozytären Reaktion nach Parasitoidenbefall wider. Die Feldlinien unterschieden sich beträchtlich in der absoluten und relativen Anzahl der verschiedenen Hämozyten, die sie als Reaktion auf den Parasitoidenbefall produzierten, selbst bei den Linien, die bei der Einkapselung sehr erfolgreich waren (Gerritsma et al., 2013). Dies unterstreicht erneut, dass der genetische Hintergrund einer Population und der kombinierte lokale Selektionsdruck zu alternativen evolutionären Reaktionen führen. Der Vergleich der Genome von resistenten und anfälligen Individuen aus verschiedenen Populationen kann die adaptive Variation in der genetischen Architektur dieses Merkmals aufzeigen.
Um die Veränderungen im Genom während der Evolution einer erhöhten Resistenz zu kartieren, haben wir eine experimentelle Evolution für Parasitoidenresistenz durchgeführt. Im Labor setzten wir eine große, überzüchtete Population Parasitoiden aus. Nur die Larven, denen es gelang, den Parasitoidenbefall zu überleben, durften zur nächsten Generation beitragen. Mit diesem Ansatz konnten wir den Grad der Resistenz von 20 auf ∼50 % der Larven, die den Parasitoidenbefall überlebten, nach nur fünf Generationen der Selektion erhöhen. Als wir die Veränderungen der Genexpression in den selektierten Populationen im Vergleich zur Genexpression in den Kontrolllinien bereits vor dem Parasitoidenbefall maßen, fanden wir mehrere Hundert Gene, die leicht unterschiedlich reguliert waren (Wertheim et al., 2011). Die Veränderungen betrafen hauptsächlich Gene, die während der Immunreaktion nicht unterschiedlich exprimiert wurden, was darauf hindeutet, dass die evolutionären Veränderungen die Immunreaktion nicht in Erwartung eines Parasitoidenangriffs voraktivierten, sondern den kanonischen Entwicklungsweg modulierten, was (auch) zu einer Steigerung der Verteidigungsfähigkeit führte. Wir wiederholten dieses Experiment und sequenzierten anschließend die Genome der ausgewählten und der Kontrollpopulationen. In den Genomen der Linien, die eine erhöhte Resistenz entwickelten, fanden wir Signaturen der Selektion in mehreren eng definierten Regionen des Genoms (Jalvingh et al., 2014). Einige dieser Regionen überschnitten sich auch mit den Regionen, die nach der Selektion auf erhöhte Resistenz eine veränderte Expression aufwiesen (Wertheim et al., 2011; Jalvingh et al., 2014). Somit kann eine schnelle und starke Selektion auf ein komplexes Merkmal wie Immunität immer noch mehrere, aber stark lokalisierte genomische Regionen betreffen.
Zukünftige Herausforderungen
Wie können wir die langfristigen evolutionären Veränderungen, wie die Übernahme neuer Gene in Geninteraktionsnetzwerken, und die kurzfristigen evolutionären Veränderungen, wie Sequenzvarianten, die eine Population durchdringen können, in Einklang bringen? Der Schlüssel dazu ist (i) die Rekonstruktion des Geninteraktionsnetzes, das komplexen Merkmalen zugrunde liegt, und (ii) die Charakterisierung der Rolle der genetischen Variation innerhalb dieser Netze. Genetische Netzwerke können sich mit neuen Genen, z. B. durch Duplikationen, erweitern, mit anderen Netzwerken oder Modulen verbunden werden, und kleine Sequenzvariationen können die Aktivität und Topologie der Netzwerke modulieren. Wenn es uns gelingt, die genetischen Netzwerke zu zerlegen und die Rolle der genetischen Varianten im Netzwerkkontext zu bewerten, können wir schließlich feststellen, wie genetische Variationen in phänotypische Variationen umgesetzt werden. Dies wird auch unser Verständnis der molekularen Grundlagen komplexer menschlicher Krankheiten und der Evolution der angeborenen und erworbenen Immunität verbessern (Cooper und Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay und Moore, 2014; Sironi et al., 2015).
Systembiologische Ansätze werden von unschätzbarem Wert sein, um die komplexen Geninteraktionsnetzwerke zu entschlüsseln. Dabei werden mathematische Modelle entwickelt, um die molekularen Mechanismen zu beschreiben, die einem Merkmal zugrunde liegen, und um die Dynamik von Gruppen interagierender Komponenten des Netzwerks vorherzusagen. Die Modelle beruhen auf molekulargenetischen und genomischen Daten. Gegenwärtig wird die Systembiologie hauptsächlich auf spezifische Merkmale von Einzellern angewandt, und dies wird als die Grenze dessen angesehen, was erreicht werden kann (Papp et al., 2011). Es ist jedoch wahrscheinlich, dass dieses Modell für die Evolution in sich sexuell fortpflanzenden mehrzelligen Organismen nicht repräsentativ ist. Anstatt darauf zu warten, dass die vereinfachten Modelle kleine Teilnetze genau widerspiegeln, müssen wir unsere Methoden entwickeln und verfeinern, um die auftauchenden Eigenschaften aus der riesigen Menge an Genomikdaten zu nutzen und zu quantifizieren. Wir können Geninteraktionsnetzwerke aus Protein-Protein- oder Transkriptkorrelationen oder Koexpressionsmatrizen ableiten (Shannon et al., 2003; Langfelder und Horvath, 2008) und diese mit Analysen natürlicher Varianten zusammenführen (Nuzhdin et al., 2012). Wir sollten diese Methoden und alternative Ansätze weiterentwickeln, um unsere Messungen an Genomikdaten voll auszuschöpfen und diese quantitativen Messungen in Netzwerkanalysen umzuwandeln. Die Genomikdaten an sich werden zwar keine vollständige Antwort auf die Frage liefern, was die Anpassungsfähigkeit des Lebens bestimmt, aber sie ermöglichen es uns, zu quantifizieren und zu beobachten, was während der Evolution auf molekularer Ebene geschieht. Wenn wir dies mit der Umweltheterogenität als Treiber von Anpassungen kombinieren und integrieren, können wir vielleicht die komplexen molekularen Mechanismen von Anpassung und Evolution aufdecken.
Erklärung zu Interessenkonflikten
Der Autor erklärt, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Ich danke Kirsten Jalvingh, Laura Salazar-Jaramillo und Sylvia Gerritsma für wertvolle Diskussionen. BW wurde von der Netherlands Organization for Scientific Research (NWO; grant 864.08.008) unterstützt.
Aggarwal, K., and Silverman, N. (2008). Positive und negative Regulierung der Immunantwort von Drosophila. BMB Rep. 41, 267-277. doi: 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Systemgenetik komplexer Merkmale in Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., and Anderson, K. V. (2004). Drosophila: die Genetik der angeborenen Immunerkennung und -reaktion. Annu. Rev. Immunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evolution der angeborenen Immunität: Hinweise von Wirbellosen über Fische zu Säugetieren. Front. Immunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., and Fitzgerald, K. A. (2014). Posttranskriptionelle Regulierung der Genexpression in der angeborenen Immunität. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., and Alder, M. N. (2006). Die Evolution des adaptiven Immunsystems. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biologie, molekular und organismisch. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Die Evolution von Genen und Genomen in der Phylogenie von Drosophila. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., und Mackay, T. F. C. (2009). Ein Transkriptionsnetzwerk im Zusammenhang mit natürlicher Variation im aggressiven Verhalten von Drosophila. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., and Doury, G. (2006). Die Fliege Drosophila subobscura: ein natürlicher Fall von Mangel an angeborener Immunität. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., and Banerjee, U. (2003). Dicker als Blut: konservierte Mechanismen in der Hämatopoese von Drosophila und Wirbeltieren. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D., and Eisen, M. B. (2004). Populationsgenetische Variation in der Genexpression ist mit phänotypischer Variation in Saccharomyces cerevisiae verbunden. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., and Allemand, R. (2009). Ecology and life history evolution of frugivorous Drosophila parasitoids. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Genomische Expressionsprogramme bei der Reaktion von Hefezellen auf Umweltveränderungen. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., and Wertheim, B. (2013). Natural variation in differentiated hemocytes is related to parasitoid resistance in Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., and Eslin, P. (2012). Strukturelle und funktionelle Charakterisierung von Pseudopodozyten, einer zotteligen Immunzelle, die von zwei Drosophila-Arten der Obscura-Gruppe produziert wird. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). Experimentelle Evolution, genetische Analyse und Genom-Resequenzierung enthüllen die Mutation, die Artemisinin-Resistenz in einer isogenen Linie von Malariaparasiten verleiht. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., and Anderson, P. (2013). Posttranskriptionelle regulatorische Netzwerke in der Immunität. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., and Wertheim, B. (2014). Genomische Veränderungen unter schneller Evolution: Selektion auf Parasitoidenresistenz. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). Die genomische Grundlage der adaptiven Evolution bei Dreistachligen Stichlingen. Nature 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Die adaptive Evolution enträtseln: Wie eine einzige Punktmutation das Protein-Koregulationsnetzwerk beeinflusst. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., und Godfray, H. C. J. (1999). Geografische Muster in der Entwicklung von Resistenz und Virulenz bei Drosophila und ihren Parasitoiden. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., und Godfray, H. C. J. (2002). Kosten der Resistenz in Insekten-Parasiten- und Insekten-Parasitoiden-Interaktionen. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., und van Alphen, J. J. M. (1995). Geografische Variation in der Verkapselungsfähigkeit von Drosophila melanogaster-Larven und Hinweise auf parasitoidenspezifische Komponenten. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., and Horvath, S. (2008). WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., and Strand, M. R. (2002). Insektenhämozyten und ihre Rolle in der Immunität. Insect Biochem. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Little, T. J. (2009). Immunität in einer variablen Welt. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Schneider, D. S. (2014). The genetics of immunity. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Vom Genotyp zum Phänotyp: Lehren aus Modellorganismen für die Humangenetik. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., and Hoffmann, J. (2007). Die Wirtsabwehr von Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., and O’Neill, L. A. J. (2005). Negative Regulierung von Toll-like-Rezeptor-vermittelten Immunantworten. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). Adaptive Evolution mehrerer Merkmale durch mehrere Mutationen an einem einzigen Gen. Science 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., and Moore, J. H. (2014). Warum Epistase wichtig ist, um die Genetik komplexer menschlicher Krankheiten anzugehen. Genome Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). Die Suche nach der fehlenden Erblichkeit komplexer Krankheiten. Nature 461, 747-753. doi: 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Multinukleierte Riesenhämozyten sind Effektorzellen in zellvermittelten Immunantworten von Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., and Williams, S. M. (eds). (2015). Epistasis. New York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., and Plasterk, R. (2003). Das Genom maßschneidern: die Macht der genetischen Ansätze. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., and Stubbs, L. (2009). Unterschiede in den Genexpressionsmustern von Mensch und Schimpanse definieren ein sich entwickelndes Netzwerk von Transkriptionsfaktoren im Gehirn. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., and McIntyre, L. M. (2012). Genotyp-Phänotyp-Kartierung in einer Post-GWAS-Welt. Trends Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J., Kim, K.-W., and Jiggins, F. M. (2009). Quantifizierung der adaptiven Evolution im Immunsystem von Drosophila. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., and Pál, C. (2011). Systembiologische Ansätze zur Vorhersage der genomischen Evolution. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., and Pittendrigh, B. R. (2004). Genomweites Transkriptionsprofil von feld- und laborselektierten Dichlordiphenyltrichlorethan (DDT)-resistenten Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistase – die wesentliche Rolle von Gen-Interaktionen in der Struktur und Evolution genetischer Systeme. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., und Siva-Jothy, M. T. (2003). Invertebrate ecological immunology. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., und Clark, A. G. (2007). Dynamische Evolution des angeborenen Immunsystems in Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., and Wertheim, B. (2014). Evolution einer zellulären Immunantwort in Drosophila: eine vergleichende phänotypische und genomische Analyse. Genome Biol. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Molekulare Netzwerke als Sensoren und Treiber für häufige menschliche Krankheiten. Nature 461, 218-223. doi: 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., und Clark, A. G. (2007). Contrasting infection strategies in generalist and specialist wasp parasitoids of Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variation in der Immunabwehr als eine Frage der Evolutionsökologie. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Evolutionäre Ökologie der Immunabwehr von Insekten. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: eine Softwareumgebung für integrierte Modelle biomolekularer Interaktionsnetzwerke. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., and Maniatis, T. (2001). NF-κB-Signalwege in der angeborenen Immunität von Säugetieren und Insekten. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., and Clerici, M. (2015). Evolutionäre Einblicke in Wirt-Pathogen-Interaktionen aus Sequenzdaten von Säugetieren. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Anpassungsgenomik: die nächste Generation. Trends Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). Die Genomsequenz des atlantischen Kabeljaus offenbart ein einzigartiges Immunsystem. Nature 477, 207-210. doi: 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Einhundert Jahre Pleiotropie: ein Rückblick. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., and Wheat, C. W. (2010). Integration von evolutionären und funktionalen Ansätzen zur Ableitung von Anpassungen an spezifischen Loci. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). Die molekulare Vielfalt der adaptiven Konvergenz. Science 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., and Tarone, A. M. (2011). Populationsbasierte Resequenzierung experimentell entwickelter Populationen offenbart die genetische Grundlage der Variation der Körpergröße bei Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., and Altenberg, L. (1996). Komplexe Anpassungen und die Evolution der Evolvierbarkeit. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., and Godfray, H. C. (2011). Funktionelle Genomik der Evolution von erhöhter Resistenz gegen Parasitismus bei Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Genomweite Genexpression als Reaktion auf Parasitoidenbefall bei Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., and Aderem, A. (2014). Systems-level analysis of innate immunity. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar