Frontiers in Genetics
Introduktion
Många egenskaper regleras och samordnas av flera gener och miljöförhållanden. I naturen utvecklas sådana egenskaper kontinuerligt under lokalt selektionstryck och neutrala processer, vilket leder till en rik mångfald av fenotypiska sorter och miljöanpassningsstrategier. Redan Dobzhansky (1964) beskrev den rika mångfalden i den levande världen som ett resultat av genetisk mångfald, miljömässig heterogenitet och anpassningar som utvecklas i samspelet mellan molekylär och organismisk biologi. De molekylära mekanismerna i evolutionen har varit särskilt svåra att karakterisera. För det första måste vi koppla genotypen till fenotypen. Denna länk består av omfattande och intrikata geninteraktionsnätverk (Ayroles et al., 2009; Lehner, 2013). För det andra måste vi identifiera de molekylära förändringar som är ansvariga för fenotypiska anpassningar. Detta är beroende av att man belyser den genetiska variationen och de genetiska förändringar som kan ske var som helst i geninteraktionsnätverken (t.ex. Edwards et al., 2009).
Den genetiska variationens manifestation i gennätverken är mycket komplex. Förändringar i enskilda gener kan påverka aktiviteten och till och med topologin i hela det genetiska nätverket (t.ex. Knight et al., 2006). Gener och geninteraktionsnätverk är ofta pleiotropa och reglerar olika egenskaper och processer, vilket innebär att förändringar i en enskild gen kan manifesteras i flera egenskaper (Stearns, 2010). Omvänt kan alleliska variationer i många gener bidra till variationen i en viss fenotypisk egenskap (Manolio et al., 2009). Vidare är epistasering genomgående, vilket innebär att alleliska variationer på flera loci kan påverka varandras fenotypiska effekter (Phillips, 2008; Mackay och Moore, 2014; Moore och Williams, 2015). Dessa överväganden om evolutionens genetiska grund är inte nya och har studerats under flera decennier (t.ex. Wagner och Altenberg, 1996). Framåtriktad och omvänd genetik har varit mycket framgångsrik när det gäller att belysa funktionerna hos enskilda gener eller mutationer för en viss egenskap (Nagy et al., 2003). Dessa tekniker är dock begränsande när det gäller att studera komplexiteten hos de molekylära interaktionsnätverk som ligger till grund för en fenotyp, eller de molekylära mekanismerna för evolutionen av komplexa egenskaper.
Den utveckling som skett inom genomiktekniken har varit ett stort lyft för vår förmåga att studera den genetiska komplexiteten hos fenotypiska egenskaper och deras evolution (Stapley et al., 2010). Genom att kombinera dessa tekniker med de klassiska genetiska metoderna kan vi bedöma den genetiska variationens funktionalitet för fenotypiska egenskaper (Storz och Wheat, 2010). De första genomiska studierna på flera modellorganismer betonade att evolutionära anpassningar, även för specifika miljöförhållanden, i allmänhet styr många gener eller loci, liksom den dynamiska regleringen av genuttrycksmönster (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). Under det följande decenniet användes genomik i många studier för att identifiera gener och proteiner som bidrog till särskilda egenskaper och ekologiska interaktioner. Till en början var de kostnader och den tid som krävdes för att sekvensera ett enskilt genom fortfarande mycket restriktiva. Next Generation Sequencing har dock gjort det möjligt att sekvensera genomet hos många fler arter och många fler individer per art. Detta är en formidabel resurs för att studera evolutionen, eftersom det för första gången gör det möjligt för oss att kartlägga förändringarna i hela genomet under evolutionen.
Genomteknik har en enorm potential att förbättra våra insikter i evolutionära processer. Jämförande metoder har tillämpats för att kartlägga förändringarna i genomsekvenserna eller geninteraktionsnätverken under långa evolutionära tidsskalor (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Metoder för experimentellt urval eller experimentell evolution, följt av antingen transkriptomik eller genomsekvensering, har använts för att kartlägga evolutionära förändringar på mycket kortare tidsskalor (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Dessa studier visade till exempel hur genduplikationer, mutationer och stark sekvensdivergens i en liten delmängd gener kan ha en djupgående inverkan på den transkriptionella aktiviteten i stora geninteraktionsnätverk och flera fenotypiska egenskaper.
I det här perspektivet presenterar jag de senaste rönen och utvecklingen när det gäller evolutionens genomiska grund, med utvecklande immunitet som en fallstudie. Immunitet är en egenskap som utvecklas snabbt, vilket gör den lämplig för att studera den genomiska grunden för evolutionära processer (Obbard et al., 2009; Sironi et al., 2015). Dessutom har molekylära nätverk inom immunitet karakteriserats ganska väl på grund av deras betydelse för människors hälsa (Schadt, 2009; Lazzaro och Schneider, 2014; Zak et al., 2014). Först sammanfattar jag mycket kortfattat vår nuvarande förståelse av de molekylära nätverken för medfödda immunsvar och de urvalsprocesser som verkar på immunsvar. Därefter beskriver jag de genomiska förändringar som är förknippade med vinst, förlust och modulering av särskilda aspekter av immunsvar i Drosophila. Slutligen föreslår jag framtida inriktningar för att studera den genetiska arkitekturen hos komplexa egenskaper och evolutionära processer.
Immunresponser
Immunsystemet består av en kombination av fysiologiska processer som agerar gemensamt i försvaret mot patogener och parasiter. Medfödd immunitet är en gammal egenskap som finns hos alla flercelliga organismer, medan ryggradsdjur även besitter förvärvad immunitet. Båda immunsystemen kombinerar cellulära och humorala komponenter: den cellulära komponenten består av specialiserade celler som ger en skyddande funktion. Detta inkluderar klasser av blodkroppar för fagocytos av mikrober, inkapsling av större främmande kroppar eller igenkänning av antigener (i förvärvad immunitet), och tarmens slemhinna med epitelceller som bildar en fysisk barriär och kan utsöndra försvarsföreningar. Den humorala komponenten består av frisättning av extracellulära faktorer som bekämpar de invaderande patogenerna, ofta från specialiserade vävnader eller cellpopulationer. Detta inkluderar frisättning av antimikrobiella peptider (AMP) från levern (eller fettkroppen hos ryggradslösa djur) och tarmepitelceller, reaktiva syrearter i fagocytiska celler och epitelceller samt antikroppar från vita blodkroppar (vid förvärvad immunitet; Lemaitre och Hoffmann, 2007; Buchmann, 2014).
Komplexa nätverk av molekylära interaktioner samordnar immunsvaret (figur 1A). Samma vägar är centrala för immunsvar från ryggradslösa djur till ryggradsdjur, vilket innebär ett starkt bevarande av kärnelementen i de molekylära nätverken i immuniteten (Silverman och Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). En mängd olika receptormolekyler kan känna igen patogener eller parasiter, till exempel baserat på patogenassocierade molekylära mönster (t.ex. lipopolysackarider som är specifika för bakteriemembran). När receptorerna väl är aktiverade inducerar de specifika signaltransduktionsvägar, såsom Toll-, Imd- och Jak/Stat-vägarna (granskad i Lemaitre och Hoffmann, 2007; Buchmann, 2014). Dessa vägar består av proteaser, kinaser, cytokiner och andra proteiner som slutligen aktiverar transkriptionsfaktorer och kofaktorer. Induktion av dessa transkriptionsfaktorer resulterar i produktion av humorala effektormolekyler (t.ex. AMP) och det kan inducera proliferation och differentiering av celler som är involverade i immuniteten. Produktionen av olika klasser av blodceller är en framträdande aspekt av den cellulära komponenten av immunsvaret, både vid medfödd och förvärvad immunitet. I dessa blodceller regleras också signaltransduktionskaskader för att framkalla cellegenskaper och proteiner som leder till att parasiten avlägsnas. För att reglera styrkan, specificiteten, tidpunkten och varaktigheten hos immunsvaret moduleras de molekylära nätverken av cytokiner, proteaser och korsverkan med andra signalvägar (Liew et al., 2005; Aggarwal och Silverman, 2008). Detta inkluderar även olika posttranskriptionella regleringsnätverk (Ivanov och Anderson, 2013; Carpenter et al., 2014).
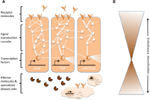
Figur 1. Schematisk representation av de genetiska nätverken i immunitet. (A) Flera sammankopplade nätverk samordnar svaren på en immunutmaning. Dessa nätverk består av proteiner (representerade av cirklar) som interagerar med varandra i en signaltransduktionskaskad för att reglera uttrycket av transkriptionsfaktorer (representerade av hexagonaler). Aktiveringen av de centrala signaltransduktionsvägarna (t.ex. IMD, Toll eller Jak/Stat, som anges med tjocka linjer mellan proteinerna) resulterar i produktion av effektormolekyler, t.ex. antimikrobiella peptider (som representeras av tårtformade symboler) och spridning och differentiering av specialiserade (blod)celler (molnformade figurer). Extracellulära och membranbundna receptormolekyler (månformade figurer) inducerar vägarna. Aktiviteten kan moduleras ytterligare av många andra proteiner som interagerar med banorna och samverkar med andra banor och genetiska nätverk (indikeras av de tunna linjerna mellan proteinerna). (B) De centrala komponenterna i de genetiska nätverken i immunitet, t.ex. transkriptionsfaktorerna och de proteiner som står i direkt kontakt med dessa transkriptionsfaktorer, är ofta starkt bevarade mellan olika stamceller. Evolutionär diversifiering finns i större utsträckning mot nätverkens periferier.
Drivkrafterna bakom de evolutionära förändringarna i immunförsvaret är de kombinerade effekterna av infektionens höga fitnesskostnader för värddjuret, kostnaderna för immunitet, den rika mångfalden av patogener och parasiter som hotar värddjuren och den dynamiska samevolutionära kapprustningen mellan värddjur och patogener (Schmid-Hempel, 2003). Någon eller alla dessa aspekter kan verka i värdens lokala miljö och leda till ett starkt selektionstryck. Typen av urval varierar dock från riktad till renande till balanserande, beroende på de kostnader och fördelar som värdpopulationen upplever i sin lokala miljö. Till exempel kan en mycket virulent patogen som invaderar ett lokalsamhälle orsaka ett selektivt svep eller riktat urval för vissa resistensalleler, eftersom endast de värdar som har dessa alleler kan bidra till de kommande generationerna. Alternativt kan en mångfaldig eller samutvecklande gemenskap av patogener driva ett frekvensberoende eller balanserande urval som gynnar upprätthållandet av genetisk variation. Utvecklingen av immunförsvaret återspeglar både denna samevolutionära dynamik med parasiten och immunsystemets fysiologiska och ekologiska kostnader (Kraaijeveld et al, 2002; Rolff och Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro och Little, 2009).
Genomisk grund för evolutionär förändring i Drosophilas immunitet
Medans det centrala maskineriet för immunresponsen är starkt bevarat kan flera komponenter i de utvidgade molekylära nätverken utvecklas snabbt eller diversifieras (figur 1B). I Drosophila har snabb evolutionär förändring rapporterats för receptorer och effektorer av immunsvar (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Dessa molekyler verkar i gränssnittet mellan värddjuret och patogenen och är därför avgörande för att värddjuret ska känna igen en invaderande organism och för att förmedla immunsvarets inriktning och antagonistiska effekter på patogenen. Samtidigt är parasiten under urval för att inte bli upptäckt, för att undvika eller mildra de antagonistiska effekterna av immunsvaret. Man förväntar sig därför en dynamik med röda drottningar för dessa molekyler vid gränssnittet och för de molekyler som modulerar immunförsvaret. Varje part försöker vinna övertaget i den antagonistiska kapprustningen, vilket leder till ömsesidiga förändringar i parternas genetiska nätverk. Diversifieringen av receptor-, modulator- och effektormolekyler åstadkoms mestadels genom genduplikationer och snabba sekvensförändringar (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Vi har studerat Drosophilas immunförsvar mot parasitoidgetingar som ett modellsystem för att förstå den genomiska grunden för evolutionära processer. Drosophila-larver är värdar för en mängd olika parasitoidarter som lägger ägg i larverna (Fleury et al., 2009). När parasitoidägget kläcks (∼2-4 dagar efter parasitoidangreppet, beroende på parasitoidart och temperatur) börjar parasitoidlarven äta på värddjuret och dödar det. Vissa arter av Drosophila har en försvarsmekanism mot parasitoider genom ett medfött immunförsvar som kallas melanotisk inkapsling. Detta immunförsvar består av cellulära och humorala komponenter som tillsammans agerar för att avskilja och döda parasitoidägget. Parasitoidangrepp utlöser immunsignalöverföringsvägar som inducerar (i) proliferation och differentiering av två klasser av hemocyter (dvs. insektsblodceller) som fäster vid parasitoidägget och vid varandra, och (ii) avsättning av melanin på parasitoidägget och den cellulära kapseln runt parasitoidägget (Lemaitre och Hoffmann, 2007). Värden måste fullfölja den fullständiga inkapslingen och melaniseringen innan parasitoidägget kläcks för att överleva parasitoidangreppet.
För genomikens era hade flera gener identifierats som var involverade i immunförsvaret mot parasitsteklar. Toll- och Jak/Stat-vägarna hade identifierats som centrala komponenter för hemocytproliferation och -differentiering, och prophenoloxidasvägen för melanisering (granskad i Brennan och Anderson, 2004). Två mikroarraystudier genomfördes sedan för att identifiera ytterligare gener som potentiellt är involverade i den melanotiska inkapslingen efter parasitoidangrepp. Detta tillvägagångssätt lyfte fram många ytterligare gener som inte tidigare hade förknippats med immunförsvaret mot parasitoider och avslöjade deras tidpunkt för verkan (Wertheim et al., 2005; Schlenke et al., 2007). Studierna avslöjade flera samordnade och funktionellt sammanhängande kluster av gener som tillfälligt upp- eller nedreglerades under en del av immunsvaret (Wertheim et al., 2005). Intressant nog visades det att virulensmekanismerna hos två parasitoidarter skiljde sig åt i hur de ingrep i det genetiska nätverket för värdresponsen: en art eliminerade den initiala aktiveringen av hela nätverket, medan en annan art riktade in sig på det sista steget i kaskaden (Schlenke et al., 2007).
Arter av Drosophila skiljer sig i hög grad åt när det gäller immunitet mot parasitoider. Vissa arter är helt mottagliga för parasitoider och detta rapporterades som en immunbrist (Eslin och Doury, 2006). En närmare granskning visade dock att parasitoidresistens inte är vanligt förekommande bland alla Drosophila-arter, utan är begränsad till några få klasser. Vi visade att i en av dessa klasser, melanogaster-undergruppen, var den evolutionära vinsten av parasitoidresistens förknippad med vinsten av en ny typ av blodcell, lamellocyter, som också är begränsad till samma klass (Salazar-Jaramillo et al., 2014). Vissa Drosophila-arter utanför melanogaster-undergruppen kan också kapsla in parasitoidägg, men de verkar ha utvecklat olika typer av blodceller för inkapslingsresponsen (Havard et al., 2012; Márkus et al., 2015). Immunförsvaret mot parasitoider har utvecklats oberoende av varandra hos olika insektstaxa, ofta med något olika mekanismer och typer av blodceller (Lavine och Strand, 2002). Evolutionen av det medfödda immunförsvaret innefattar således tillägg av nya komponenter eller ”moduler”. Detta väcker frågan om hur genomet förändras under förvärvet av en ny modul.
Komparativ genomik visade att trots förvärvet av en ny typ av blodcell i melanogaster-undergruppen är de gener som är kända för lamellocytdifferentiering i stort sett bevarade över hela fylogenin. Även arter som inte producerar lamellocyter som svar på parasitoidangrepp har dessa gener som krävs för differentiering av lamellocyter. Dessutom uppvisar dessa gener lite divergens eller signaturer av selektion, medan man skulle kunna förvänta sig det för gener som fått en ny funktion. Detta tyder på att de befintliga signaltransduktionsvägarna för differentiering av hemocyter moduleras av det omgivande geninteraktionsnätverket för att producera en ny typ av blodcell i melanogasterundergruppen. Denna samodifiering av den befintliga centrala hemocytproliferationsvägen uppnås sannolikt genom att andra eller nya komponenter läggs till i geninteraktionsnätverket (Salazar-Jaramillo et al., 2014). Vi identifierade flera nya gener som uppstod runt tiden för förvärvet av lamellocyter och som uttrycks differentiellt under immunsvaret mot parasitoider, inklusive receptormolekyler och proteaser av serintyp (Salazar-Jaramillo et al., 2014). Vi antar att särskilt endopeptidaserna av serintyp kan spela en avgörande roll i denna expansion av geninteraktionsnätverket. Ett betydande antal av dessa molekyler uppstod vid tidpunkten för förvärvet av lamellocyter, de uttrycks vid rätt tidpunkt i immunsvaret och de visar starka signaturer av positivt urval (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
Också inom en enda art, D. melanogaster, uppvisar immunsvaret stor genetisk variation. Fältpopulationer som samlats in från hela Europa visar betydande skillnader i förmågan att framgångsrikt kapsla in parasitoidägg (Kraaijeveld och van Alphen, 1995; Kraaijeveld och Godfray, 1999; Gerritsma et al., 2013). Tydligen skiljer sig kostnaderna och fördelarna med ett starkt immunförsvar åt geografiskt, vilket leder till modulering och differentiering av samanpassade genetiska nätverk. Detta återspeglades också i det hemocytära svaret efter parasitangrepp. Fältlinjerna varierade avsevärt i det absoluta och relativa antalet av de olika hemocyter som de producerade som svar på parasitoidangrepp, även bland de linjer som var mycket framgångsrika i inkapsling (Gerritsma et al., 2013). Detta understryker återigen att den genetiska bakgrunden i en population och det kombinerade lokala selektionstrycket leder till alternativa evolutionära svar. Att jämföra genomet hos resistenta och mottagliga individer från flera populationer kan avslöja den adaptiva variationen i den genetiska arkitekturen för denna egenskap.
För att kartlägga förändringarna i genomet under evolutionen av ökad resistens genomförde vi experimentell evolution för parasitoidresistens. I laboratoriet utsatte vi en stor outbred population för parasitoider. Endast de larver som lyckades överleva parasitoidangrepp fick bidra till nästa generation. Med detta tillvägagångssätt ökade vi resistensnivån från 20 till ∼50 % av de larver som överlevde parasitoidangrepp efter endast fem generationers urval. När vi mätte förändringar i genuttrycket i de selekterade populationerna, jämfört med genuttrycket i kontrollinjerna, även före parasitoidangrepp, fann vi flera hundra gener som var något differentiellt reglerade (Wertheim et al., 2011). Förändringarna gällde mestadels gener som inte var differentiellt uttryckta under immunsvaret, vilket tyder på att de evolutionära förändringarna inte föraktiverade immunsvaret i avvaktan på parasitoidangrepp, utan att de modulerade den kanoniska utvecklingsvägen, vilket (också) ledde till en ökning av dess försvarsförmåga. Vi upprepade detta experiment och sekvenserade sedan genomet hos de utvalda och kontrollpopulationerna. I genomet hos de linjer som utvecklade ökad motståndskraft hittade vi signaturer av selektion på flera snävt definierade regioner av genomet (Jalvingh et al., 2014). Några av dessa regioner överlappade också med de regioner som visade förändrat uttryck efter selektion för ökad resistens (Wertheim et al., 2011; Jalvingh et al., 2014). Således kan en snabb och stark selektiv svepning på en komplex egenskap som immunitet fortfarande påverka flera, men mycket lokaliserade, genomiska regioner.
Framtida utmaningar
Hur ska vi förena de långsiktiga evolutionära förändringarna, såsom förvärv av nya gener i geninteraktionsnätverk, och de kortsiktiga evolutionära förändringarna, såsom sekvensvarianter som kan svepas genom en population? Nyckeln till detta är att i) rekonstruera det geninteraktionsnätverk som ligger till grund för komplexa egenskaper och ii) karaktärisera den genetiska variationens roll i dessa nätverk. Genetiska nätverk kan expandera med nya gener genom t.ex. duplikationer, bli sammankopplade med andra nätverk eller moduler, och små sekvensvariationer kan modulera nätverkens aktivitet och topologi. Om vi kan dela upp de genetiska nätverken och bedöma den roll som genetiska varianter spelar i ett nätverkssammanhang kommer detta så småningom att göra det möjligt för oss att fastställa hur genetisk variation omvandlas till fenotypisk variation. Detta kommer också att förbättra vår förståelse av den molekylära grunden för komplexa mänskliga sjukdomar och evolutionen av medfödd och förvärvad immunitet (Cooper och Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay och Moore, 2014; Sironi et al., 2015).
Systembiologiska metoder kommer att vara ovärderliga för att reda ut de komplexa geninteraktionsnätverken. Där utvecklas matematiska modeller för att beskriva de molekylära mekanismer som ligger till grund för en egenskap och för att förutsäga dynamiken hos grupper av interagerande komponenter i nätverket. Modellerna bygger på molekylärgenetiska och genomiska data. För närvarande tillämpas systembiologi främst på specifika egenskaper hos encelliga organismer, och detta anses vara gränsen för vad som kan uppnås (Papp et al., 2011). Det är dock troligt att denna modell kanske inte är representativ för evolutionen i sexuellt reproducerande flercelliga organismer. I stället för att vänta på att de förenklade modellerna ska återspegla små delnätverk på ett korrekt sätt måste vi utveckla och förfina våra metoder för att utnyttja och kvantifiera de framväxande egenskaperna från den stora mängden genomikdata. Vi kan härleda geninteraktionsnätverk från protein-protein- eller transkriptkorrelation eller samuttrycksmatriser (Shannon et al., 2003; Langfelder och Horvath, 2008) och slå samman dessa med analyser av naturliga varianter (Nuzhdin et al., 2012). Vi bör vidareutveckla dessa metoder och alternativa tillvägagångssätt för att fullt ut utnyttja våra mätningar av genomikdata och för att omvandla dessa kvantitativa mätningar till nätverksanalyser. Även om genomikdata i sig inte kommer att ge det fullständiga svaret på vad som bestämmer livets anpassningsförmåga, gör de det möjligt för oss att kvantifiera och observera vad som händer på molekylär nivå under evolutionen. När vi kombinerar och integrerar detta med miljöheterogenitet som drivkraft för anpassningar kan vi kanske avslöja de komplexa molekylära mekanismerna för anpassning och evolution.
Intressekonfliktförklaring
Författaren förklarar att forskningen utfördes i avsaknad av kommersiella eller finansiella relationer som skulle kunna tolkas som en potentiell intressekonflikt.
Acknowledgments
Jag tackar Kirsten Jalvingh, Laura Salazar-Jaramillo och Sylvia Gerritsma för värdefulla diskussioner. BW fick stöd från Nederländernas organisation för vetenskaplig forskning (NWO; bidrag 864.08.008).
Aggarwal, K., and Silverman, N. (2008). Positiv och negativ reglering av Drosophilas immunsvar. BMB Rep. 41, 267-277. doi: 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Systemgenetik för komplexa egenskaper hos Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., and Anderson, K. V. (2004). Drosophila: genetiken för det medfödda immunförsvarets erkännande och respons. Annu. Rev. Immunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evolution av den medfödda immuniteten: ledtrådar från ryggradslösa djur via fiskar till däggdjur. Front. Immunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J. och Fitzgerald, K. A. (2014). Post-transkriptionell reglering av genuttryck vid medfödd immunitet. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., and Alder, M. N. (2006). Evolutionen av adaptiva immunsystem. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biologi, molekylär och organismisk. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Utveckling av gener och genomer i Drosophila-fylogenin. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F. och Mackay, T. F. C. (2009). Ett transkriptionellt nätverk som är förknippat med naturlig variation i Drosophila aggressivt beteende. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., and Doury, G. (2006). Flugan Drosophila subobscura: ett naturligt fall av bristande medfödd immunitet. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V. och Banerjee, U. (2003). Tjockare än blod: bevarade mekanismer i Drosophila och vertebraternas hematopoiesis. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D. och Eisen, M. B. (2004). Populationsgenetisk variation i genuttryck är associerad med fenotypisk variation i Saccharomyces cerevisiae. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N. och Allemand, R. (2009). Ekologi och livshistorisk utveckling hos frugivorösa Drosophila-parasitoider. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Genomiska uttrycksprogram i jästcellernas svar på miljöförändringar. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L. och Wertheim, B. (2013). Naturlig variation i differentierade hemocyter är relaterad till parasitoidresistens hos Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G. och Eslin, P. (2012). Strukturell och funktionell karakterisering av pseudopodocyten, en lurvig immuncell som produceras av två Drosophila-arter i obscura-gruppen. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). Experimentell evolution, genetisk analys och omsekvensering av genomet avslöjar den mutation som ger artemisininresistens i en isogen linje av malariaparasiter. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., and Anderson, P. (2013). Post-transkriptionella regleringsnätverk i immunitet. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V. och Wertheim, B. (2014). Genomiska förändringar under snabb evolution: urval för parasitoidresistens. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). Den genomiska grunden för adaptiv evolution hos trepine sticklebacks. Nature 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Unraveling adaptive evolution: how a single point mutation affects the protein coregulation network. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and Godfray, H. C. J. (1999). Geografiska mönster i utvecklingen av resistens och virulens hos Drosophila och dess parasitoider. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., and Godfray, H. C. J. (2002). Kostnader för resistens i interaktioner mellan insekter-parasiter och insekter-parasitoider. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and van Alphen, J. J. M. (1995). Geografisk variation i inkapslingsförmåga hos larver av Drosophila melanogaster och bevis för parasitoidspecifika komponenter. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., and Horvath, S. (2008). WGCNA: ett R-paket för analys av viktade korrelationsnätverk. BMC Bioinformatics 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., and Strand, M. R. (2002). Insekts hemocyter och deras roll i immuniteten. Insect Biochem. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Little, T. J. (2009). Immunitet i en föränderlig värld. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Schneider, D. S. (2014). Immunitetens genetik. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Genotyp till fenotyp: lärdomar från modellorganismer för humangenetik. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., and Hoffmann, J. (2007). Värdförsvaret hos Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K. och O’Neill, L. A. J. (2005). Negativ reglering av Toll-like receptor-medierade immunsvar. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). Adaptiv utveckling av flera egenskaper genom flera mutationer i en enda gen. Science 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., and Moore, J. H. (2014). Varför epistasis är viktigt för att ta itu med komplex mänsklig sjukdomsgenetik. Genome Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). Att hitta den saknade arvbarheten för komplexa sjukdomar. Nature 461, 747-753. doi: 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Multinukleerade jättehemocyter är effektorceller i cellmedierade immunsvar hos Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., and Williams, S. M. (eds). (2015). Epistasis. New York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S. och Plasterk, R. (2003). Att skräddarsy genomet: kraften i genetiska metoder. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E. och Stubbs, L. (2009). Skillnader i genuttrycksmönster hos människor och schimpanser definierar ett utvecklande nätverk av transkriptionsfaktorer i hjärnan. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L. och McIntyre, L. M. (2012). Kartläggning av genotyp och fenotyp i en värld efter GWAS. Trends Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J. J., Kim, K.-W. och Jiggins, F. M. (2009). Kvantifiering av adaptiv evolution i Drosophilas immunsystem. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., and Pál, C. (2011). Systembiologiska metoder för att förutsäga genomisk evolution. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E. och Pittendrigh, B. R. (2004). Transkriptionsprofil över hela genomet hos fält- och laboratorieselekterad diklordifenyltrichloretan (DDT)-resistent Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistasis-the essential role of gene interactions in the structure and evolution of genetic systems. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., and Siva-Jothy, M. T. (2003). Ekologisk immunologi hos ryggradslösa djur. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D. och Clark, A. G. (2007). Dynamisk utveckling av det medfödda immunsystemet i Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T. och Wertheim, B. (2014). Evolutionen av ett cellulärt immunsvar hos Drosophila: en fenotypisk och genomisk komparativ analys. Genome Biol. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Molekylära nätverk som sensorer och drivkrafter för vanliga mänskliga sjukdomar. Nature 461, 218-223. doi: 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S. och Clark, A. G. (2007). Kontrasterande infektionsstrategier hos generalistiska och specialiserade getingparasitoider hos Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variation i immunförsvaret som en fråga om evolutionär ekologi. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Evolutionär ekologi för insekters immunförsvar. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: en mjukvarumiljö för integrerade modeller av biomolekylära interaktionsnätverk. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., and Maniatis, T. (2001). NF-κB-signalvägar i däggdjurs och insekters medfödda immunitet. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D. och Clerici, M. (2015). Evolutionära insikter om värd-patogeninteraktioner från sekvensdata från däggdjur. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Anpassningsgenomik: nästa generation. Trends Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). Genomsekvensen hos torsk avslöjar ett unikt immunsystem. Nature 477, 207-210. doi: 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Hundra år av pleiotropi: en tillbakablick. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., and Wheat, C. W. (2010). Integrering av evolutionära och funktionella metoder för att härleda anpassning vid specifika loci. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). Den molekylära mångfalden av adaptiv konvergens. Science 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., and Tarone, A. M. (2011). Populationsbaserad resekvensering av experimentellt utvecklade populationer avslöjar den genetiska grunden för variation i kroppsstorlek hos Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., and Altenberg, L. (1996). Komplexa anpassningar och evolutionen av utvecklingsbarhet. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M. och Godfray, H. C. (2011). Funktionell genomik av evolutionen av ökad resistens mot parasitism hos Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Genomövergripande genuttryck som svar på parasitoidangrepp hos Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., and Aderem, A. (2014). Analys på systemnivå av den medfödda immuniteten. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar