Frontiers in Genetics
Introduzione
Molti tratti sono regolati e coordinati da più geni e condizioni ambientali. In natura, questi tratti si evolvono continuamente sotto pressioni di selezione locale e processi neutri, portando ad una ricca diversità di varietà fenotipiche e strategie ambientali. Già Dobzhansky (1964) ha descritto la ricca diversità nel mondo vivente come il risultato della diversità genetica, dell’eterogeneità ambientale e degli adattamenti che si evolvono nell’interazione tra biologia molecolare e organismica. I meccanismi molecolari nell’evoluzione sono stati particolarmente difficili da caratterizzare. In primo luogo, dobbiamo collegare il genotipo al fenotipo. Questo collegamento consiste in reti di interazione genica estese e intricate (Ayroles et al., 2009; Lehner, 2013). In secondo luogo, dobbiamo identificare i cambiamenti molecolari responsabili degli adattamenti fenotipici. Questo dipende dal chiarimento della variazione genetica e dei cambiamenti genetici che possono verificarsi ovunque nelle reti di interazione genica (ad esempio, Edwards et al., 2009).
La manifestazione della variazione genetica nelle reti geniche è molto complessa. I cambiamenti in singoli geni possono influenzare l’attività e persino la topologia dell’intera rete genetica (ad esempio, Knight et al., 2006). I geni e le reti di interazione genica sono spesso pleiotropici e regolano vari tratti e processi, il che implica che i cambiamenti in un singolo gene possono manifestarsi in diversi tratti (Stearns, 2010). Al contrario, le variazioni alleliche in molti geni possono contribuire alla variazione di un particolare tratto fenotipico (Manolio et al., 2009). Inoltre, l’epistasi è pervasiva, il che implica che le variazioni alleliche a più loci possono influenzare gli effetti fenotipici l’una dell’altra (Phillips, 2008; Mackay e Moore, 2014; Moore e Williams, 2015). Queste considerazioni sulla base genetica dell’evoluzione non sono nuove, e sono state studiate per diversi decenni (ad esempio, Wagner e Altenberg, 1996). La genetica diretta e inversa ha avuto un grande successo nel chiarire le funzioni di singoli geni o mutazioni per un particolare tratto (Nagy et al., 2003). Queste tecniche, tuttavia, sono limitanti quando si studia la complessità delle reti di interazione molecolare alla base di un fenotipo, o i meccanismi molecolari dell’evoluzione di tratti complessi.
Gli sviluppi della tecnologia genomica sono stati una spinta importante per la nostra capacità di studiare la complessità genetica dei tratti fenotipici e la loro evoluzione (Stapley et al., 2010). La combinazione di queste tecniche con gli approcci genetici classici ci permette di valutare la funzionalità della variazione genetica per i tratti fenotipici (Storz e Wheat, 2010). I primi studi genomici su diversi organismi modello hanno sottolineato che gli adattamenti evolutivi, anche per specifiche condizioni ambientali, generalmente governano molti geni o loci, così come la regolazione dinamica dei modelli di espressione genica (Gasch et al., 2000; Fay et al., 2004; Pedra et al., 2004). Nel decennio successivo, molti studi hanno utilizzato la genomica per identificare i geni e le proteine che contribuivano a particolari tratti e interazioni ecologiche. Inizialmente, i costi e il tempo richiesti per il sequenziamento di un singolo genoma erano ancora molto restrittivi. Il Next Generation Sequencing, tuttavia, ha reso possibile sequenziare i genomi di molte più specie e molti più individui per specie. Questa è una risorsa formidabile per studiare l’evoluzione, poiché ci permette, per la prima volta, di mappare i cambiamenti nell’intero genoma durante l’evoluzione.
La tecnologia genomica ha un enorme potenziale per migliorare la nostra comprensione dei processi evolutivi. Gli approcci comparativi sono stati applicati per mappare i cambiamenti nelle sequenze del genoma o nelle reti di interazione genica su lunghe scale temporali evolutive (Drosophila 12 Genomes Consortium et al., 2007; Nowick et al., 2009; Jones et al., 2012). Gli approcci di selezione sperimentale o di evoluzione sperimentale, seguiti dalla trascrittomica o dal sequenziamento del genoma, sono stati utilizzati per mappare i cambiamenti evolutivi su scale temporali molto più brevi (Hunt et al., 2010; Turner et al., 2011; Wertheim et al., 2011; Tenaillon et al., 2012; Linnen et al., 2013; Jalvingh et al., 2014). Questi studi hanno mostrato, per esempio, come duplicazioni geniche, mutazioni e forti divergenze di sequenza in un piccolo sottoinsieme di geni possono avere un profondo impatto sull’attività trascrizionale di grandi reti di interazione genica e su molteplici tratti fenotipici.
In questa prospettiva, presento recenti risultati e sviluppi sulla base genomica dell’evoluzione, utilizzando l’immunità in evoluzione come caso di studio. L’immunità è un tratto che si evolve rapidamente, rendendolo adatto allo studio delle basi genomiche dei processi evolutivi (Obbard et al., 2009; Sironi et al., 2015). Inoltre, le reti molecolari nell’immunità sono state abbastanza ben caratterizzate a causa della loro importanza per la salute umana (Schadt, 2009; Lazzaro e Schneider, 2014; Zak et al., 2014). In primo luogo, riassumo molto brevemente la nostra attuale comprensione delle reti molecolari delle risposte immunitarie innate e dei processi di selezione che agiscono sulle risposte immunitarie. Poi, descrivo i cambiamenti genomici, associati con il guadagno, la perdita e la modulazione di particolari aspetti delle risposte immunitarie in Drosophila. Infine, propongo direzioni future per studiare l’architettura genetica di tratti complessi e processi evolutivi.
Risposte immunitarie
Il sistema immunitario consiste in una combinazione di processi fisiologici che agiscono congiuntamente nella difesa contro patogeni e parassiti. L’immunità innata è un tratto antico che si trova in tutti gli organismi pluricellulari, mentre i vertebrati possiedono anche l’immunità acquisita. Entrambi i sistemi immunitari combinano componenti cellulari e umorali: la componente cellulare comprende cellule specializzate che forniscono una funzione protettiva. Questo include, classi di cellule del sangue per la fagocitosi di microbi, incapsulamento di corpi estranei più grandi o riconoscimento di antigeni (nell’immunità acquisita), e il rivestimento dell’intestino con cellule epiteliali che formano una barriera fisica e possono secernere composti difensivi. La componente umorale consiste nel rilascio di fattori extracellulari che combattono gli agenti patogeni invasori, spesso da tessuti specializzati o popolazioni di cellule. Questo include il rilascio di peptidi antimicrobici (AMPs) dal fegato (o dal corpo grasso negli invertebrati) e dalle cellule epiteliali dell’intestino, specie reattive dell’ossigeno nelle cellule fagocitarie ed epiteliali, e anticorpi dai globuli bianchi (nell’immunità acquisita; Lemaitre e Hoffmann, 2007; Buchmann, 2014).
Reti complesse di interazioni molecolari coordinano le risposte immunitarie (Figura 1A). Gli stessi percorsi sono centrali per le risposte immunitarie dagli invertebrati ai vertebrati, implicando una forte conservazione degli elementi centrali delle reti molecolari nell’immunità (Silverman e Maniatis, 2001; Evans et al., 2003; Buchmann, 2014). Una varietà di molecole recettoriali può riconoscere gli agenti patogeni o i parassiti, per esempio, sulla base di modelli molecolari associati al patogeno (ad esempio, lipopolisaccaridi specifici delle membrane batteriche). Una volta che i recettori sono attivati, inducono specifiche vie di trasduzione del segnale, come le vie Toll, Imd e Jak/Stat (riviste in Lemaitre e Hoffmann, 2007; Buchmann, 2014). Queste vie consistono di proteasi, chinasi, citochine e altre proteine che alla fine attivano fattori di trascrizione e cofattori. L’induzione di questi fattori di trascrizione si traduce nella produzione di molecole effettrici umorali (ad esempio, AMPs) e può indurre la proliferazione e la differenziazione delle cellule coinvolte nell’immunità. La produzione di diverse classi di cellule del sangue è un aspetto importante della componente cellulare della risposta immunitaria, sia nell’immunità innata che in quella acquisita. In queste cellule del sangue, le cascate di trasduzione del segnale sono anche regolate per indurre le proprietà delle cellule e le proteine che effettuano la clearance del parassita. Per regolare la forza, la specificità, la tempistica e la durata delle risposte immunitarie, le reti molecolari sono modulate da citochine, proteasi e cross-talk con altre vie di segnalazione (Liew et al., 2005; Aggarwal e Silverman, 2008). Questo include anche diverse reti di regolazione post-trascrizionale (Ivanov e Anderson, 2013; Carpenter et al., 2014).
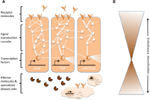
Figura 1. Rappresentazione schematica delle reti genetiche nell’immunità. (A) Diverse reti interconnesse coordinano le risposte a una sfida immunitaria. Queste reti consistono di proteine (rappresentate da cerchi) che interagiscono tra loro in una cascata di trasduzione del segnale per regolare l’espressione di fattori di trascrizione (rappresentati da esagoni). L’attivazione delle vie principali di trasduzione del segnale (per esempio, IMD, Toll, o Jak/Stat, indicate da linee spesse tra le proteine) ha come risultato la produzione di molecole effettrici, come i peptidi antimicrobici (rappresentati da simboli a forma di torta) e la proliferazione e differenziazione di cellule specializzate (sangue) (figure a forma di nuvola). Le molecole recettoriali extracellulari e legate alla membrana (figure a forma di luna) inducono i percorsi. L’attività può essere ulteriormente modulata da molte altre proteine che interagiscono con i pathways e cross-talk con altri pathways e reti genetiche (indicate dalle linee sottili tra le proteine). (B) I componenti centrali delle reti genetiche nell’immunità, per esempio, i fattori di trascrizione e le proteine in contatto diretto con questi fattori di trascrizione, sono spesso fortemente conservati attraverso i phyla. La diversificazione evolutiva si trova più estesamente verso le periferie delle reti.
I motori dei cambiamenti evolutivi nelle risposte immunitarie sono gli effetti combinati degli alti costi di fitness dell’infezione sugli ospiti, i costi dell’immunità, la ricca diversità di patogeni e parassiti che minacciano gli ospiti, e le gare dinamiche di co-evoluzione tra ospiti e patogeni (Schmid-Hempel, 2003). Uno qualsiasi o tutti questi aspetti possono operare nell’ambiente locale dell’ospite e portare a forti pressioni di selezione. Il tipo di selezione, tuttavia, varia da direzionale a purificante a equilibrante, a seconda dei costi e dei benefici che la popolazione ospite sperimenta nel suo ambiente locale. Per esempio, un patogeno altamente virulento che invade una comunità locale può causare una spazzata selettiva o una selezione direzionale per particolari alleli di resistenza, poiché solo gli ospiti con questi alleli possono contribuire alle generazioni successive. In alternativa, una comunità di patogeni diversi o in co-evoluzione può guidare una selezione dipendente dalla frequenza o di bilanciamento, favorendo il mantenimento della variazione genetica. L’evoluzione delle risposte immunitarie riflette sia queste dinamiche coevolutive con il parassita, sia i costi fisiologici ed ecologici del sistema immunitario (Kraaijeveld et al, 2002; Rolff e Siva-Jothy, 2003; Schmid-Hempel, 2005; Lazzaro e Little, 2009).
Base genomica del cambiamento evolutivo nell’immunità di Drosophila
Mentre il meccanismo centrale delle risposte immunitarie è fortemente conservato, diversi componenti delle reti molecolari estese possono evolvere rapidamente o diversificarsi (Figura 1B). In Drosophila, un rapido cambiamento evolutivo è stato riportato per i recettori e gli effettori della risposta immunitaria (Sackton et al., 2007; Obbard et al., 2009; Salazar-Jaramillo et al., 2014). Queste molecole operano all’interfaccia tra l’ospite e il patogeno, e sono quindi cruciali per il riconoscimento da parte dell’ospite di un organismo invasore, e per mediare il targeting e gli effetti antagonisti della risposta immunitaria sul patogeno. Allo stesso tempo, il parassita è sotto selezione per passare inosservato, per evitare o mitigare gli effetti antagonisti della risposta immunitaria. Pertanto, ci si aspetta una dinamica da Regina Rossa per queste molecole all’interfaccia e quelle che modulano le risposte immunitarie. Ogni parte sta cercando di ottenere il sopravvento nella corsa agli armamenti antagonisti, guidando reciprocamente le alterazioni nelle reti genetiche delle parti. La diversificazione nelle molecole recettore, modulatore ed effettore è per lo più realizzata da duplicazioni geniche e rapidi cambiamenti di sequenza (Drosophila 12 Genomes Consortium et al., 2007; Sackton et al., 2007; Salazar-Jaramillo et al., 2014).
Abbiamo studiato la risposta immunitaria della Drosophila contro le vespe parassitoidi come sistema modello per comprendere la base genomica dei processi evolutivi. Le larve di Drosophila ospitano una varietà di specie di parassitoidi che depongono un uovo in queste larve (Fleury et al., 2009). Una volta che l’uovo del parassitoide si schiude (∼2-4 giorni dopo l’attacco del parassitoide, a seconda della specie di parassitoide e della temperatura), la larva del parassitoide inizia a nutrirsi dell’ospite e lo uccide. Alcune specie di Drosophila hanno un meccanismo di difesa contro i parassitoidi attraverso una risposta immunitaria innata, chiamata incapsulamento melanotico. Questa risposta immunitaria consiste in componenti cellulari e umorali che agiscono congiuntamente per sequestrare e uccidere l’uovo del parassitoide. L’attacco del parassitoide innesca vie di trasduzione del segnale immunitario che inducono (i) la proliferazione e la differenziazione di due classi di emociti (cioè le cellule del sangue degli insetti) che aderiscono all’uovo del parassitoide e tra loro, e (ii) la deposizione di melanina sull’uovo del parassitoide e sulla capsula cellulare attorno all’uovo del parassitoide (Lemaitre e Hoffmann, 2007). L’ospite deve completare l’incapsulamento completo e la melanizzazione prima che l’uovo del parassitoide si schiuda per sopravvivere all’infestazione del parassitoide.
Prima dell’era della genomica, erano stati identificati diversi geni coinvolti nella risposta immunitaria contro le vespe parassitoidi. Le vie Toll e Jak/Stat erano state identificate come componenti centrali della proliferazione e differenziazione degli emociti, e la via della profenolossidasi per la melanizzazione (rivista in Brennan e Anderson, 2004). Sono stati quindi condotti due studi di microarray per identificare ulteriori geni potenzialmente coinvolti nell’incapsulamento melanotico dopo l’attacco dei parassitoidi. Questo approccio ha evidenziato molti geni aggiuntivi che non erano stati precedentemente associati alla risposta immunitaria contro i parassitoidi, e ha rivelato i loro tempi di azione (Wertheim et al., 2005; Schlenke et al., 2007). Gli studi hanno rivelato diversi gruppi coordinati e funzionalmente coerenti di geni che sono stati temporaneamente up- o down-regolati durante una parte della risposta immunitaria (Wertheim et al., 2005). È interessante notare che i meccanismi di virulenza di due specie di parassitoidi differivano nel modo in cui interferivano nella rete genetica delle risposte dell’ospite: una specie eliminava l’attivazione iniziale dell’intera rete, mentre un’altra specie mirava al passo finale della cascata (Schlenke et al., 2007).
Le specie di Drosophila differiscono ampiamente nell’immunità contro i parassitoidi. Alcune specie sono completamente suscettibili ai parassitoidi e questo è stato riportato come un deficit immunitario (Eslin e Doury, 2006). Un’ispezione più approfondita, tuttavia, ha rivelato che la resistenza ai parassitoidi non è comunemente condivisa tra tutte le specie di Drosophila, ma è limitata ad alcuni cladi. Abbiamo dimostrato che in uno di questi cladi, il sottogruppo melanogaster, il guadagno evolutivo della resistenza ai parassitoidi era associato al guadagno di un nuovo tipo di cellula sanguigna, i lamellociti, che è anche limitato allo stesso clade (Salazar-Jaramillo et al., 2014). Alcune specie di Drosophila al di fuori del sottogruppo melanogaster possono anche incapsulare le uova dei parassitoidi, ma sembrano aver evoluto diversi tipi di cellule del sangue per la risposta di incapsulamento (Havard et al., 2012; Márkus et al., 2015). La risposta immunitaria contro i parassitoidi si è evoluta indipendentemente in vari taxa di insetti, spesso con meccanismi e tipi di cellule ematiche leggermente diversi (Lavine e Strand, 2002). Così, l’evoluzione del sistema immunitario innato include l’aggiunta di nuovi componenti o “moduli”. Questo solleva la questione di come il genoma cambia durante l’acquisizione di un nuovo modulo.
La genomica comparativa ha rivelato che, nonostante l’acquisizione di un nuovo tipo di cellule del sangue nel sottogruppo melanogaster, i geni che sono noti per la differenziazione dei lamellociti sono ampiamente conservati in tutta la filogenesi. Anche le specie che non producono lamellociti in risposta all’attacco dei parassitoidi possiedono questi geni che sono richiesti per la differenziazione dei lamellociti. Inoltre, questi geni mostrano poca divergenza o firme di selezione, mentre ciò sarebbe previsto per i geni che hanno ottenuto una nuova funzione. Questo indica che le vie di trasduzione del segnale esistenti per la differenziazione degli emociti sono modulate dalla rete di interazione genica circostante per produrre un nuovo tipo di cellule del sangue nel sottogruppo melanogaster. Questa cooptazione della via di proliferazione degli emociti esistente è probabilmente ottenuta aggiungendo altri o nuovi componenti alla rete di interazione genica (Salazar-Jaramillo et al., 2014). Abbiamo identificato diversi nuovi geni che sono sorti intorno al momento dell’acquisizione dei lamellociti e sono differenzialmente espressi durante la risposta immunitaria contro i parassitoidi, comprese le molecole recettrici e le proteasi di tipo serina (Salazar-Jaramillo et al., 2014). Noi ipotizziamo che soprattutto le endopeptidasi di tipo serina possano giocare un ruolo cruciale in questa espansione della rete di interazione genica. Un numero sostanziale di queste molecole è sorto al momento dell’acquisizione dei lamellociti, sono espresse al momento giusto nella risposta immunitaria, e mostrano forti firme di selezione positiva (Wertheim et al., 2005; Salazar-Jaramillo et al., 2014).
Anche all’interno di una singola specie, D. melanogaster, le risposte immunitarie mostrano grandi variazioni genetiche. Le popolazioni di campo raccolte in tutta Europa mostrano differenze sostanziali nella capacità di incapsulare con successo le uova dei parassitoidi (Kraaijeveld e van Alphen, 1995; Kraaijeveld e Godfray, 1999; Gerritsma et al., 2013). Apparentemente, i costi e i benefici di una forte difesa immunitaria differiscono geograficamente, portando alla modulazione e alla differenziazione delle reti genetiche co-adattate. Questo si riflette anche nella risposta emocitica dopo l’attacco dei parassitoidi. Le linee di campo variavano considerevolmente nel numero assoluto e relativo dei diversi emociti che producevano in risposta all’attacco dei parassitoidi, anche tra le linee che avevano grande successo nell’incapsulamento (Gerritsma et al., 2013). Questo sottolinea nuovamente che il background genetico di una popolazione e le pressioni di selezione locali combinate portano a risposte evolutive alternative. Confrontare i genomi di individui resistenti e suscettibili di diverse popolazioni può rivelare la variazione adattativa nell’architettura genetica di questo tratto.
Per mappare i cambiamenti nel genoma durante l’evoluzione di una maggiore resistenza, abbiamo condotto un’evoluzione sperimentale per la resistenza dei parassitoidi. In laboratorio abbiamo esposto una grande popolazione di outbred ai parassitoidi. Solo le larve che sono riuscite a sopravvivere all’attacco dei parassitoidi hanno potuto contribuire alla generazione successiva. Con questo approccio, abbiamo aumentato il livello di resistenza dal 20 al ∼50% delle larve che sopravvivono all’attacco dei parassitoidi dopo solo cinque generazioni di selezione. Quando abbiamo misurato i cambiamenti nell’espressione genica nelle popolazioni selezionate, rispetto all’espressione genica nelle linee di controllo, anche prima dell’attacco dei parassitoidi, abbiamo trovato diverse centinaia di geni che erano regolati in modo leggermente diverso (Wertheim et al., 2011). I cambiamenti hanno coinvolto per lo più geni che non erano differenzialmente espressi durante la risposta immunitaria, indicando che i cambiamenti evolutivi non hanno pre-attivato la risposta immunitaria in previsione dell’attacco dei parassitoidi, ma hanno modulato il percorso di sviluppo canonico, che (anche) porta ad un aumento delle sue capacità difensive. Abbiamo ripetuto questo esperimento, e poi sequenziato i genomi delle popolazioni selezionate e di controllo. Nei genomi delle linee che hanno evoluto una maggiore resistenza, abbiamo trovato firme di selezione su più regioni strettamente definite del genoma (Jalvingh et al., 2014). Alcune di queste regioni si sono anche sovrapposte alle regioni che hanno mostrato un’espressione modificata dopo la selezione per una maggiore resistenza (Wertheim et al., 2011; Jalvingh et al., 2014). Così, una rapida e forte spazzata selettiva su un tratto complesso come l’immunità può ancora influenzare più regioni genomiche, ma altamente localizzate.
Sfide future
Come faremo a conciliare i cambiamenti evolutivi a lungo termine, come l’acquisizione di nuovi geni nelle reti di interazione genica, e i cambiamenti evolutivi a breve termine, come le varianti di sequenza che possono essere spazzate attraverso una popolazione? La chiave per questo è (i) ricostruire la rete di interazione genica sottostante i tratti complessi e (ii) caratterizzare il ruolo della variazione genetica all’interno di queste reti. Le reti genetiche possono espandersi con nuovi geni attraverso, per esempio, duplicazioni, diventare interconnesse con altre reti o moduli, e piccole variazioni di sequenza possono modulare l’attività e la topologia delle reti. Se possiamo scomporre le reti genetiche e valutare il ruolo delle varianti genetiche in un contesto di rete, questo ci permetterà alla fine di determinare come la variazione genetica si traduce in variazione fenotipica. Questo migliorerà anche la nostra comprensione della base molecolare delle malattie umane complesse e l’evoluzione dell’immunità innata e acquisita (Cooper e Alder, 2006; Manolio et al., 2009; Star et al., 2011; Mackay e Moore, 2014; Sironi et al., 2015).
Gli approcci biologici sistemici saranno preziosi per svelare le complesse reti di interazione genica. Lì, vengono sviluppati modelli matematici per descrivere i meccanismi molecolari alla base di un tratto e per prevedere le dinamiche di gruppi di componenti interagenti della rete. I modelli sono basati su dati di genetica molecolare e genomica. Al momento, la biologia dei sistemi è per lo più applicata a tratti specifici in organismi unicellulari, e questo è considerato il limite a ciò che può essere raggiunto (Papp et al., 2011). È probabile, tuttavia, che questo modello possa non essere rappresentativo per l’evoluzione negli organismi multicellulari che si riproducono sessualmente. Piuttosto che aspettare che i modelli semplificati riflettano accuratamente le piccole sottoreti, dobbiamo sviluppare e raffinare i nostri metodi per utilizzare e quantificare le proprietà emergenti dalla grande quantità di dati genomici. Possiamo dedurre le reti di interazione genica dalla correlazione proteina-proteina o trascrizione o dalle matrici di co-espressione (Shannon et al., 2003; Langfelder e Horvath, 2008), e fonderle con le analisi delle varianti naturali (Nuzhdin et al., 2012). Dovremmo sviluppare ulteriormente questi metodi e approcci alternativi per sfruttare appieno le nostre misure sui dati genomici, e per convertire queste misure quantitative in analisi di rete. Mentre i dati genomici di per sé non forniranno la risposta completa a ciò che determina la capacità di adattamento della vita, ci permettono di quantificare e osservare ciò che accade a livello molecolare durante l’evoluzione. Quando combiniamo e integriamo questo con l’eterogeneità ambientale come driver degli adattamenti, potremmo essere in grado di rivelare i complessi meccanismi molecolari di adattamento ed evoluzione.
Dichiarazione di conflitto di interessi
L’autore dichiara che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimenti
Ringrazio Kirsten Jalvingh, Laura Salazar-Jaramillo, e Sylvia Gerritsma per preziose discussioni. BW è stato sostenuto da un finanziamento della Netherlands Organization for Scientific Research (NWO; grant 864.08.008).
Aggarwal, K., and Silverman, N. (2008). Regolazione positiva e negativa della risposta immunitaria Drosophila. BMB Rep. 41, 267-277. doi: 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Genetica dei sistemi di tratti complessi in Drosophila melanogaster. Nat. Genet. 41, 299-307. doi: 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., and Anderson, K. V. (2004). Drosophila: la genetica del riconoscimento immunitario innato e la risposta. Annu. Rev. Immunol. 22, 457-483. doi: 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Evoluzione dell’immunità innata: indizi da invertebrati via pesce ai mammiferi. Fronte. Immunol. 5:459. doi: 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., e Fitzgerald, K. A. (2014). Regolazione post-trascrizionale dell’espressione genica nell’immunità innata. Nat. Rev. Immunol. 14, 361-376. doi: 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., and Alder, M. N. (2006). L’evoluzione dei sistemi immunitari adattativi. Cell 124, 815-822. doi: 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). Biologia, molecolare e organismica. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Evoluzione dei geni e dei genomi sulla filogenesi di Drosophila. Nature 450, 203-218. doi: 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., e Mackay, T. F. C. (2009). Una rete trascrizionale associata alla variazione naturale nel comportamento aggressivo di Drosophila. Genome Biol. 10, R76. doi: 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., e Doury, G. (2006). La mosca Drosophila subobscura: un caso naturale di deficit di immunità innata. Dev. Comp. Immunol. 30, 977-983. doi: 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., e Banerjee, U. (2003). Più spesso del sangue: meccanismi conservati nell’ematopoiesi di Drosophila e dei vertebrati. Dev. Cell 5, 673-690. doi: 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D., and Eisen, M. B. (2004). La variazione genetica di popolazione nell’espressione genica è associata alla variazione fenotipica in Saccharomyces cerevisiae. Genome Biol. 5, R26. doi: 10.1186/gb-2004-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., e Allemand, R. (2009). Ecologia ed evoluzione della storia di vita dei parassitoidi frugivori di Drosophila. Adv. Parasitol. 70, 3-44. doi: 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. Programmi di espressione genomica nella risposta delle cellule di lievito ai cambiamenti ambientali. Mol. Biol. Cell 11, 4241-4257. doi: 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., and Wertheim, B. (2013). Variazione naturale in emociti differenziati è legato alla resistenza parassitoide in Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi: 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., e Eslin, P. (2012). Caratterizzazione strutturale e funzionale di pseudopodocyte, una cellula immunitaria ispida prodotta da due specie di Drosophila del gruppo obscura. Dev. Comp. Immunol. 36, 323-331. doi: 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. Evoluzione sperimentale, analisi genetica e ri-sequenziamento del genoma rivelano la mutazione che conferisce resistenza artemisinina in una stirpe isogenica di parassiti della malaria. BMC Genomics 11:499. doi: 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., e Anderson, P. (2013). Reti di regolazione post-trascrizionale nell’immunità. Immunol. Rev. 253, 253-272. doi: 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., e Wertheim, B. (2014). Cambiamenti genomici sotto rapida evoluzione: selezione per la resistenza parassitoide. Proc. Biol. Sci. 281, 20132303. doi: 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). La base genomica dell’evoluzione adattativa in spinarelli threespine. Natura 484, 55-61. doi: 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. Svelare l’evoluzione adattativa: come una singola mutazione puntiforme influenza la rete di coregolazione delle proteine. Nat. Genet. 38, 1015. doi: 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and Godfray, H. C. J. (1999). Modelli geografici nell’evoluzione della resistenza e della virulenza in Drosophila e nei suoi parassitoidi. Am. Nat. 153, S61-S74. doi: 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., and Godfray, H. C. J. (2002). Costi della resistenza nelle interazioni insetto-parassita e insetto-parassitoide. Parasitology 125(Suppl.), S71-S82. doi: 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., and van Alphen, J. J. M. (1995). Variazione geografica nella capacità di incapsulamento delle larve di Drosophila melanogaster e prove per componenti specifiche del parassitoide. Evol. Ecol. 9, 10-17. doi: 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., and Horvath, S. (2008). WGCNA: un pacchetto R per l’analisi delle reti di correlazione pesata. BMC Bioinformatica 9:559. doi: 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., and Strand, M. R. (2002). Emociti di insetti e il loro ruolo nell’immunità. Insect Biochem. Mol. Biol. 32, 1295-1309. doi: 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Little, T. J. (2009). Immunità in un mondo variabile. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi: 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., e Schneider, D. S. (2014). La genetica dell’immunità. G3 (Bethesda) 4, 943-945. doi: 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Genotype to phenotype: lezioni da organismi modello per la genetica umana. Nat. Rev. Genet. 14, 168-178. doi: 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., e Hoffmann, J. (2007). La difesa dell’ospite di Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi: 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., and O’Neill, L. A. J. (2005). Regolazione negativa delle risposte immunitarie mediate dai recettori Toll-like. Nat. Rev. Immunol. 5, 446-458. doi: 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. Evoluzione adattativa di tratti multipli attraverso mutazioni multiple a un singolo gene. Scienza 339, 1312-1316. doi: 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., e Moore, J. H. (2014). Perché epistasi è importante per affrontare la genetica complessa malattia umana. Genome Med. 6, 42. doi: 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. Trovare l’ereditabilità mancante delle malattie complesse. Natura 461, 747-753. doi: 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Emociti giganti multinucleati sono cellule effettrici nelle risposte immunitarie cellulo-mediate di Drosophila. J. Innate Immun. doi: 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., e Williams, S. M. (eds). (2015). Epistasi. New York, NY: Springer. doi: 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., e Plasterk, R. (2003). Personalizzare il genoma: il potere degli approcci genetici. Nat. Genet. 33(Suppl.), 276-284. doi: 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., e Stubbs, L. (2009). Le differenze nei modelli di espressione genica umana e scimpanzé definire una rete in evoluzione dei fattori di trascrizione nel cervello. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi: 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., e McIntyre, L. M. (2012). Mappatura genotipo-fenotipo in un mondo post-GWAS. Trends Genet. 28, 421-426. doi: 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J., Kim, K.-W., e Jiggins, F. M. (2009). Quantificare l’evoluzione adattativa nel sistema immunitario Drosophila. PLoS Genet. 5:e1000698. doi: 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., e Pál, C. (2011). Approcci di biologia dei sistemi per prevedere l’evoluzione genomica. Nat. Rev. Genet. 12, 591-602. doi: 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., e Pittendrigh, B. R. (2004). Genome-wide profilo di trascrizione di campo e laboratorio selezionati diclorodifeniltricloroetano (DDT) resistente Drosophila. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi: 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistasi: il ruolo essenziale delle interazioni geniche nella struttura e nell’evoluzione dei sistemi genetici. Nat. Rev. Genet. 9, 855-867. doi: 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., and Siva-Jothy, M. T. (2003). Immunologia ecologica degli invertebrati. Science 301, 472-475. doi: 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., and Clark, A. G. (2007). Evoluzione dinamica del sistema immunitario innato in Drosophila. Nat. Genet. 39, 1461-1468. doi: 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., e Wertheim, B. (2014). Evoluzione di una risposta immunitaria cellulare in Drosophila: un’analisi comparativa fenotipica e genomica. Genoma Biol. Evol. 6, 273-289. doi: 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Reti molecolari come sensori e driver di malattie umane comuni. Natura 461, 218-223. doi: 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., e Clark, A. G. (2007). Strategie di infezione contrastanti in parassitoidi vespa generalista e specialista di Drosophila melanogaster. PLoS Pathog. 3:e158. doi: 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). Variazione nella difesa immunitaria come una questione di ecologia evolutiva. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi: 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Ecologia evolutiva delle difese immunitarie degli insetti. Annu. Rev. Entomol. 50, 529-551. doi: 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape: un ambiente software per modelli integrati di reti di interazione biomolecolare. Genome Res. 13, 2498-2504. doi: 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., e Maniatis, T. (2001). NF-κB vie di segnalazione nei mammiferi e l’immunità innata insetto. Genes Dev. 15, 2321-2342. doi: 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., e Clerici, M. (2015). Approfondimenti evolutivi nelle interazioni ospite-patogeno dai dati di sequenza dei mammiferi. Nat. Rev. Genet. 16, 224-236. doi: 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Genomica dell’adattamento: la prossima generazione. Trends Ecol. Evol. 25, 705-712. doi: 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). La sequenza del genoma del merluzzo atlantico rivela un sistema immunitario unico. Natura 477, 207-210. doi: 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Cento anni di pleiotropia: una retrospettiva. Genetics 186, 767-773. doi: 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., e Wheat, C. W. (2010). Integrazione di approcci evolutivi e funzionali per dedurre l’adattamento a loci specifici. Evolution 64, 2489-2509. doi: 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. La diversità molecolare della convergenza adattativa. Scienza 335, 457-461. doi: 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., e Tarone, A. M. (2011). Risequenziamento basato sulla popolazione di popolazioni evolute sperimentalmente rivela la base genetica della variazione delle dimensioni del corpo in Drosophila melanogaster. PLoS Genet. 7:e1001336. doi: 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., e Altenberg, L. (1996). Adattamenti complessi e l’evoluzione dell’evolvibilità. Evolution 50, 967-976. doi: 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., e Godfray, H. C. (2011). Genomica funzionale dell’evoluzione di una maggiore resistenza al parassitismo in Drosophila. Mol. Ecol. 20, 932-949. doi: 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. Genome-wide espressione genica in risposta all’attacco parassitoide in Drosophila. Genome Biol. 11, R94. doi: 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., e Aderem, A. (2014). Analisi a livello di sistemi di immunità innata. Annu. Rev. Immunol. 32, 547-577. doi: 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar