Frontiers in Genetics
Introduction
De nombreux traits sont régulés et coordonnés par de multiples gènes et conditions environnementales. Dans la nature, de tels traits évoluent continuellement sous des pressions de sélection locales et des processus neutres, conduisant à une riche diversité de variétés phénotypiques et de stratégies d’adaptation à l’environnement. Déjà, Dobzhansky (1964) décrivait la riche diversité du monde vivant comme le résultat de la diversité génétique, de l’hétérogénéité environnementale et des adaptations qui évoluent à l’interaction entre la biologie moléculaire et la biologie organique. Les mécanismes moléculaires de l’évolution ont été particulièrement difficiles à caractériser. Tout d’abord, nous devons établir un lien entre le génotype et le phénotype. Ce lien est constitué de réseaux d’interactions génétiques étendus et complexes (Ayroles et al., 2009 ; Lehner, 2013). Deuxièmement, nous devons identifier les changements moléculaires responsables des adaptations phénotypiques. Cela dépend de l’élucidation de la variation génétique et des changements génétiques qui peuvent se produire n’importe où dans les réseaux d’interactions génétiques (par exemple, Edwards et al., 2009).
La manifestation de la variation génétique dans les réseaux de gènes est très complexe. Les changements dans des gènes uniques peuvent affecter l’activité et même la topologie de l’ensemble du réseau génétique (par exemple, Knight et al., 2006). Les gènes et les réseaux d’interactions génétiques sont souvent pléiotropes et régulent divers traits et processus, ce qui implique que les changements dans un seul gène peuvent se manifester dans plusieurs traits (Stearns, 2010). Inversement, les variations alléliques dans de nombreux gènes peuvent contribuer à la variation d’un trait phénotypique particulier (Manolio et al., 2009). En outre, l’épistasie est omniprésente, ce qui implique que les variations alléliques de plusieurs loci peuvent affecter les effets phénotypiques les uns des autres (Phillips, 2008 ; Mackay et Moore, 2014 ; Moore et Williams, 2015). Ces considérations sur la base génétique de l’évolution ne sont pas nouvelles, et sont étudiées depuis plusieurs décennies (par exemple, Wagner et Altenberg, 1996). La génétique directe et la génétique inverse ont été très efficaces pour élucider les fonctions de gènes uniques ou de mutations pour un caractère particulier (Nagy et al., 2003). Ces techniques sont cependant limitées lorsqu’il s’agit d’étudier la complexité des réseaux d’interactions moléculaires sous-jacents à un phénotype, ou les mécanismes moléculaires de l’évolution de traits complexes.
Les développements de la technologie génomique ont été un coup de pouce majeur pour notre capacité à étudier les complexités génétiques des traits phénotypiques et leur évolution (Stapley et al., 2010). La combinaison de ces techniques avec les approches classiques de la génétique nous permet d’évaluer la fonctionnalité de la variation génétique pour les traits phénotypiques (Storz et Wheat, 2010). Les premières études génomiques sur plusieurs organismes modèles ont souligné que les adaptations évolutives, même pour des conditions environnementales spécifiques, régissent généralement de nombreux gènes ou loci, ainsi que la régulation dynamique des modèles d’expression génétique (Gasch et al., 2000 ; Fay et al., 2004 ; Pedra et al., 2004). Au cours de la décennie suivante, de nombreuses études ont utilisé la génomique pour identifier les gènes et les protéines qui contribuaient à des traits particuliers et à des interactions écologiques. Au départ, les coûts et le temps nécessaires au séquençage d’un seul génome étaient encore très contraignants. Le séquençage de nouvelle génération a toutefois permis de séquencer les génomes de beaucoup plus d’espèces, et de beaucoup plus d’individus par espèce. C’est une ressource formidable pour étudier l’évolution, car elle nous permet, pour la première fois, de cartographier les changements sur l’ensemble du génome au cours de l’évolution.
La technologie génomique a un énorme potentiel pour améliorer notre compréhension des processus évolutifs. Des approches comparatives ont été appliquées pour cartographier les changements dans les séquences du génome ou les réseaux d’interaction des gènes à des échelles de temps évolutives longues (Drosophila 12 Genomes Consortium et al., 2007 ; Nowick et al., 2009 ; Jones et al., 2012). Les approches de sélection expérimentale ou d’évolution expérimentale, suivies par la transcriptomique ou le séquençage du génome, ont été utilisées pour cartographier les changements évolutifs à des échelles de temps beaucoup plus courtes (Hunt et al., 2010 ; Turner et al., 2011 ; Wertheim et al., 2011 ; Tenaillon et al., 2012 ; Linnen et al., 2013 ; Jalvingh et al., 2014). Ces études ont montré, par exemple, comment les duplications de gènes, les mutations et les fortes divergences de séquences dans un petit sous-ensemble de gènes peuvent avoir un impact profond sur l’activité transcriptionnelle de grands réseaux d’interactions génétiques et de multiples traits phénotypiques.
Dans cette perspective, je présente les découvertes et les développements récents sur la base génomique de l’évolution, en utilisant l’immunité évolutive comme étude de cas. L’immunité est un trait qui évolue rapidement, ce qui la rend propice à l’étude de la base génomique des processus évolutifs (Obbard et al., 2009 ; Sironi et al., 2015). En outre, les réseaux moléculaires dans l’immunité ont été assez bien caractérisés en raison de leur importance pour la santé humaine (Schadt, 2009 ; Lazzaro et Schneider, 2014 ; Zak et al., 2014). Tout d’abord, je résume très brièvement notre compréhension actuelle sur les réseaux moléculaires des réponses immunitaires innées et les processus de sélection qui agissent sur les réponses immunitaires. Ensuite, je décris les changements génomiques, associés au gain, à la perte et à la modulation d’aspects particuliers des réponses immunitaires chez la drosophile. Enfin, je propose des orientations futures pour étudier l’architecture génétique des traits complexes et des processus évolutifs.
Réponses immunitaires
Le système immunitaire consiste en une combinaison de processus physiologiques qui agissent conjointement dans la défense contre les pathogènes et les parasites. L’immunité innée est un trait ancien que l’on retrouve chez tous les organismes pluricellulaires, tandis que les vertébrés possèdent également une immunité acquise. Les deux systèmes immunitaires combinent des composantes cellulaires et humorales : la composante cellulaire comprend des cellules spécialisées qui assurent une fonction de protection. Il s’agit notamment de classes de cellules sanguines pour la phagocytose des microbes, l’encapsulation de corps étrangers plus gros ou la reconnaissance d’antigènes (dans l’immunité acquise), et la paroi de l’intestin avec des cellules épithéliales qui forment une barrière physique et peuvent sécréter des composés défensifs. La composante humorale consiste en la libération de facteurs extracellulaires qui combattent les agents pathogènes envahissants, souvent par des tissus ou des populations cellulaires spécialisés. Cela inclut la libération de peptides antimicrobiens (AMP) par le foie (ou le corps gras chez les invertébrés) et les cellules épithéliales de l’intestin, d’espèces réactives de l’oxygène dans les cellules phagocytaires et épithéliales, et d’anticorps provenant des globules blancs (dans l’immunité acquise ; Lemaitre et Hoffmann, 2007 ; Buchmann, 2014).
Des réseaux complexes d’interactions moléculaires coordonnent les réponses immunitaires (figure 1A). Les mêmes voies sont centrales aux réponses immunitaires des invertébrés aux vertébrés, ce qui implique une forte conservation des éléments centraux des réseaux moléculaires dans l’immunité (Silverman et Maniatis, 2001 ; Evans et al., 2003 ; Buchmann, 2014). Diverses molécules réceptrices peuvent reconnaître des agents pathogènes ou des parasites, par exemple sur la base de motifs moléculaires associés aux agents pathogènes (par exemple, les lipopolysaccharides spécifiques des membranes bactériennes). Une fois les récepteurs activés, ils induisent des voies de transduction du signal spécifiques, telles que les voies Toll, Imd et Jak/Stat (examinées dans Lemaitre et Hoffmann, 2007 ; Buchmann, 2014). Ces voies comprennent des protéases, des kinases, des cytokines et d’autres protéines qui finissent par activer des facteurs de transcription et des cofacteurs. L’induction de ces facteurs de transcription entraîne la production de molécules effectrices humorales (par exemple, les AMP) et elle peut induire la prolifération et la différenciation des cellules impliquées dans l’immunité. La production de différentes classes de cellules sanguines est un aspect important de la composante cellulaire de la réponse immunitaire, tant dans l’immunité innée que dans l’immunité acquise. Dans ces cellules sanguines, des cascades de transduction de signaux sont également régulées pour induire des propriétés cellulaires et des protéines qui assurent la clairance du parasite. Pour réguler la force, la spécificité, le moment et la durée des réponses immunitaires, les réseaux moléculaires sont modulés par des cytokines, des protéases et des échanges avec d’autres voies de signalisation (Liew et al., 2005 ; Aggarwal et Silverman, 2008). Cela inclut également divers réseaux de régulation post-transcriptionnelle (Ivanov et Anderson, 2013 ; Carpenter et al., 2014).
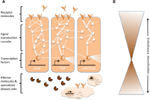
Figure 1. Représentation schématique des réseaux génétiques dans l’immunité. (A) Plusieurs réseaux interconnectés coordonnent les réponses à un défi immunitaire. Ces réseaux sont constitués de protéines (représentées par des cercles) qui interagissent entre elles dans une cascade de transduction du signal pour réguler l’expression des facteurs de transcription (représentés par des hexagones). L’activation des principales voies de transduction du signal (par exemple, IMD, Toll ou Jak/Stat, indiquées par des lignes épaisses entre les protéines) entraîne la production de molécules effectrices, telles que des peptides antimicrobiens (représentés par des symboles en forme de tarte), ainsi que la prolifération et la différenciation de cellules spécialisées (sang) (figures en forme de nuage). Les molécules réceptrices extracellulaires et membranaires (figures en forme de lune) induisent les voies. L’activité peut être modulée par de nombreuses autres protéines qui interagissent avec les voies et interagissent avec d’autres voies et réseaux génétiques (indiqués par les lignes fines entre les protéines). (B) Les composants centraux des réseaux génétiques dans l’immunité, par exemple les facteurs de transcription et les protéines en contact direct avec ces facteurs de transcription, sont souvent fortement conservés à travers les phyla. La diversification évolutive est plus importante vers la périphérie des réseaux.
Les moteurs des changements évolutifs dans les réponses immunitaires sont les effets combinés des coûts d’adaptation élevés de l’infection sur les hôtes, les coûts de l’immunité, la riche diversité des pathogènes et des parasites qui menacent les hôtes, et les courses aux armements dynamiques co-évolutives entre les hôtes et les pathogènes (Schmid-Hempel, 2003). L’un ou l’autre de ces aspects, ou tous, peuvent agir dans l’environnement local de l’hôte et entraîner de fortes pressions de sélection. Le type de sélection, cependant, varie de directionnel à purificateur à équilibrant, selon les coûts et les avantages que la population hôte subit dans son environnement local. Par exemple, un agent pathogène très virulent qui envahit une communauté locale peut provoquer un balayage sélectif ou une sélection directionnelle pour des allèles de résistance particuliers, car seuls les hôtes possédant ces allèles peuvent contribuer aux générations suivantes. Par ailleurs, une communauté d’agents pathogènes diversifiée ou en co-évolution peut entraîner une sélection dépendante de la fréquence ou équilibrante, favorisant le maintien de la variation génétique. L’évolution des réponses immunitaires reflète à la fois cette dynamique co-évolutive avec le parasite et les coûts physiologiques et écologiques du système immunitaire (Kraaijeveld et al, 2002 ; Rolff et Siva-Jothy, 2003 ; Schmid-Hempel, 2005 ; Lazzaro et Little, 2009).
Base génomique du changement évolutif dans l’immunité de la drosophile
Alors que la machinerie centrale des réponses immunitaires est fortement conservée, plusieurs composants des réseaux moléculaires étendus peuvent évoluer rapidement ou se diversifier (figure 1B). Chez la drosophile, des changements évolutifs rapides ont été signalés pour les récepteurs et les effecteurs de la réponse immunitaire (Sackton et al., 2007 ; Obbard et al., 2009 ; Salazar-Jaramillo et al., 2014). Ces molécules opèrent à l’interface entre l’hôte et le pathogène, et sont donc cruciales pour la reconnaissance par l’hôte d’un organisme envahisseur, et pour la médiation du ciblage et des effets antagonistes de la réponse immunitaire sur le pathogène. En même temps, le parasite est sélectionné pour ne pas être détecté, afin d’éviter ou d’atténuer les effets antagonistes de la réponse immunitaire. On s’attend donc à une dynamique de la Reine Rouge pour ces molécules à l’interface et celles qui modulent les réponses immunitaires. Chaque partie tente de prendre le dessus dans cette course aux armements antagoniste, entraînant réciproquement des modifications dans les réseaux génétiques des parties. La diversification des molécules réceptrices, modulatrices et effectrices est principalement accomplie par des duplications de gènes et des changements de séquence rapides (Drosophila 12 Genomes Consortium et al., 2007 ; Sackton et al., 2007 ; Salazar-Jaramillo et al., 2014).
Nous avons étudié la réponse immunitaire de la drosophile contre les guêpes parasitoïdes comme système modèle pour comprendre la base génomique des processus évolutifs. Les larves de drosophile sont l’hôte d’une variété d’espèces de parasitoïdes qui pondent un œuf dans ces larves (Fleury et al., 2009). Une fois l’œuf de parasitoïde éclos (∼2-4 jours après l’attaque du parasitoïde, selon l’espèce de parasitoïde, et la température), la larve de parasitoïde commence à se nourrir de l’hôte et le tue. Certaines espèces de drosophiles possèdent un mécanisme de défense contre les parasitoïdes par une réponse immunitaire innée, appelée encapsulation mélanotique. Cette réponse immunitaire est constituée de composants cellulaires et humoraux qui agissent conjointement pour séquestrer et tuer l’œuf de parasitoïde. L’attaque du parasitoïde déclenche des voies de transduction du signal immunitaire qui induisent (i) la prolifération et la différenciation de deux classes d’hémocytes (c’est-à-dire les cellules sanguines des insectes) qui adhèrent à l’œuf du parasitoïde et les unes aux autres, et (ii) le dépôt de mélanine sur l’œuf du parasitoïde et la capsule cellulaire autour de l’œuf du parasitoïde (Lemaitre et Hoffmann, 2007). L’hôte doit achever l’encapsulation et la mélanisation complètes avant l’éclosion de l’œuf de parasitoïde pour survivre à l’infestation par le parasitoïde.
Avant l’ère de la génomique, plusieurs gènes avaient été identifiés comme étant impliqués dans la réponse immunitaire contre les guêpes parasitoïdes. Les voies Toll et Jak/Stat avaient été identifiées comme des composantes centrales de la prolifération et de la différenciation des hémocytes, et la voie de la prophénoloxydase pour la mélanisation (revue dans Brennan et Anderson, 2004). Deux études de microréseaux ont ensuite été menées pour identifier d’autres gènes potentiellement impliqués dans l’encapsulation mélanotique après l’attaque des parasitoïdes. Cette approche a mis en évidence de nombreux gènes supplémentaires qui n’avaient pas été précédemment associés à la réponse immunitaire contre les parasitoïdes, et a révélé leur moment d’action (Wertheim et al., 2005 ; Schlenke et al., 2007). Les études ont révélé plusieurs groupes de gènes coordonnés et fonctionnellement cohérents qui étaient temporairement régulés à la hausse ou à la baisse pendant une partie de la réponse immunitaire (Wertheim et al., 2005). De façon intéressante, il a été montré que les mécanismes de virulence de deux espèces de parasitoïdes différaient dans la façon dont ils interféraient dans le réseau génétique des réponses des hôtes : une espèce a éliminé l’activation initiale de l’ensemble du réseau, tandis qu’une autre espèce a ciblé la dernière étape de la cascade (Schlenke et al., 2007).
Les espèces de drosophiles diffèrent largement dans l’immunité contre les parasitoïdes. Certaines espèces sont complètement sensibles aux parasitoïdes et cela a été signalé comme une déficience immunitaire (Eslin et Doury, 2006). Un examen plus approfondi a toutefois révélé que la résistance aux parasitoïdes n’est pas partagée par toutes les espèces de drosophiles, mais qu’elle est limitée à quelques clades. Nous avons montré que dans l’un de ces clades, le sous-groupe melanogaster, le gain évolutif de la résistance aux parasitoïdes était associé au gain d’un nouveau type de cellule sanguine, les lamellocytes, qui est également limité au même clade (Salazar-Jaramillo et al., 2014). Certaines espèces de drosophiles en dehors du sous-groupe melanogaster peuvent également encapsuler les œufs de parasitoïdes, mais elles semblent avoir évolué vers différents types de cellules sanguines pour la réponse d’encapsulation (Havard et al., 2012 ; Márkus et al., 2015). La réponse immunitaire contre les parasitoïdes a évolué indépendamment chez divers taxons d’insectes, souvent avec des mécanismes et des types de cellules sanguines légèrement différents (Lavine et Strand, 2002). Ainsi, l’évolution du système immunitaire inné comprend l’ajout de nouveaux composants ou « modules ». Cela soulève la question de savoir comment le génome change pendant l’acquisition d’un nouveau module.
La génomique comparative a révélé que, malgré le gain d’un nouveau type de cellule sanguine dans le sous-groupe melanogaster, les gènes connus pour la différenciation des lamellocytes sont largement conservés à travers toute la phylogénie. Les espèces qui ne produisent pas de lamellocytes en réponse à l’attaque des parasitoïdes possèdent également ces gènes nécessaires à la différenciation des lamellocytes. De plus, ces gènes montrent peu de divergence ou de signatures de sélection, alors que l’on pourrait s’y attendre pour des gènes ayant obtenu une nouvelle fonction. Cela indique que les voies de transduction du signal existantes pour la différenciation des hémocytes sont modulées par le réseau d’interactions génétiques environnant pour produire un nouveau type de cellule sanguine dans le sous-groupe melanogaster. Cette cooptation de la voie principale existante de la prolifération des hémocytes est probablement réalisée en ajoutant d’autres ou de nouveaux composants au réseau d’interactions génétiques (Salazar-Jaramillo et al., 2014). Nous avons identifié plusieurs nouveaux gènes qui sont apparus à peu près au moment de l’acquisition des lamellocytes et qui sont exprimés de manière différentielle pendant la réponse immunitaire contre les parasitoïdes, notamment des molécules réceptrices et des protéases de type sérine (Salazar-Jaramillo et al., 2014). Nous supposons que les endopeptidases de type sérine en particulier peuvent jouer un rôle crucial dans cette expansion du réseau d’interactions génétiques. Un nombre substantiel de ces molécules est apparu au moment de l’acquisition des lamellocytes, elles sont exprimées au bon moment dans la réponse immunitaire, et elles montrent de fortes signatures de sélection positive (Wertheim et al., 2005 ; Salazar-Jaramillo et al., 2014).
Au sein d’une même espèce, D. melanogaster, les réponses immunitaires montrent une grande variation génétique. Les populations de terrain collectées à travers l’Europe montrent des différences substantielles dans la capacité à encapsuler avec succès les œufs de parasitoïdes (Kraaijeveld et van Alphen, 1995 ; Kraaijeveld et Godfray, 1999 ; Gerritsma et al., 2013). Apparemment, les coûts et les avantages d’une forte défense immunitaire diffèrent géographiquement, ce qui entraîne une modulation et une différenciation des réseaux génétiques co-adaptés. Cela s’est également reflété dans la réponse hémocytaire après une attaque de parasitoïdes. Les lignées de terrain varient considérablement dans les nombres absolus et relatifs des différents hémocytes qu’elles produisent en réponse à l’attaque des parasitoïdes, même parmi les lignées qui ont très bien réussi l’encapsulation (Gerritsma et al., 2013). Cela souligne à nouveau que le fond génétique d’une population et les pressions de sélection locales combinées conduisent à des réponses évolutives alternatives. La comparaison des génomes des individus résistants et sensibles de plusieurs populations peut révéler la variation adaptative de l’architecture génétique de ce trait.
Pour cartographier les changements dans le génome pendant l’évolution de la résistance accrue, nous avons mené une évolution expérimentale pour la résistance aux parasitoïdes. Dans le laboratoire, nous avons exposé une grande population consanguine à des parasitoïdes. Seules les larves qui ont réussi à survivre à l’attaque des parasitoïdes ont été autorisées à contribuer à la génération suivante. Avec cette approche, nous avons augmenté le niveau de résistance de 20 à ∼50% des larves survivant à l’attaque des parasitoïdes après seulement cinq générations de sélection. Lorsque nous avons mesuré les changements dans l’expression des gènes dans les populations sélectionnées, par rapport à l’expression des gènes dans les lignées témoins, même avant l’attaque des parasitoïdes, nous avons trouvé plusieurs centaines de gènes qui étaient légèrement régulés de manière différentielle (Wertheim et al., 2011). Ces changements concernaient principalement des gènes qui n’étaient pas exprimés de manière différentielle pendant la réponse immunitaire, ce qui indique que les changements évolutifs n’ont pas préactivé la réponse immunitaire en prévision de l’attaque des parasitoïdes, mais qu’ils ont modulé la voie de développement canonique, ce qui a (également) conduit à une augmentation de ses capacités défensives. Nous avons répété cette expérience, puis nous avons séquencé les génomes des populations sélectionnées et des populations témoins. Dans les génomes des lignées qui ont développé une résistance accrue, nous avons trouvé des signatures de sélection sur plusieurs régions étroitement définies du génome (Jalvingh et al., 2014). Certaines de ces régions se chevauchent également avec les régions dont l’expression a changé après la sélection pour une résistance accrue (Wertheim et al., 2011 ; Jalvingh et al., 2014). Ainsi, un balayage sélectif rapide et fort sur un trait complexe comme l’immunité peut encore affecter des régions génomiques multiples, mais très localisées.
Défis futurs
Comment allons-nous concilier les changements évolutifs à long terme, comme l’acquisition de nouveaux gènes dans les réseaux d’interactions génétiques, et les changements évolutifs à court terme, comme les variants de séquence qui peuvent être balayés dans une population ? La clé est de (i) reconstruire le réseau d’interactions génétiques qui sous-tend les traits complexes, et (ii) de caractériser le rôle de la variation génétique au sein de ces réseaux. Les réseaux génétiques peuvent s’étendre avec de nouveaux gènes par le biais, par exemple, de duplications, devenir interconnectés avec d’autres réseaux ou modules, et de petites variations de séquence peuvent moduler l’activité et la topologie des réseaux. Si nous pouvons décomposer les réseaux génétiques et évaluer le rôle des variantes génétiques dans un contexte de réseau, cela nous permettra éventuellement de déterminer comment la variation génétique se traduit en variation phénotypique. Cela améliorera également notre compréhension de la base moléculaire des maladies humaines complexes et de l’évolution de l’immunité innée et acquise (Cooper et Alder, 2006 ; Manolio et al., 2009 ; Star et al., 2011 ; Mackay et Moore, 2014 ; Sironi et al., 2015).
Les approches biologiques des systèmes seront précieuses pour démêler les réseaux complexes d’interactions génétiques. Là, des modèles mathématiques sont développés pour décrire les mécanismes moléculaires qui sous-tendent un trait et pour prédire la dynamique des groupes de composants en interaction du réseau. Les modèles sont basés sur des données de génétique moléculaire et de génomique. À l’heure actuelle, la biologie systémique est surtout appliquée à des traits spécifiques d’organismes unicellulaires, ce qui est considéré comme la limite de ce qui peut être réalisé (Papp et al., 2011). Il est toutefois probable que ce modèle ne soit pas représentatif de l’évolution des organismes multicellulaires à reproduction sexuée. Plutôt que d’attendre que les modèles simplifiés reflètent avec précision les petits sous-réseaux, nous devons développer et affiner nos méthodes pour utiliser et quantifier les propriétés émergentes à partir de la grande quantité de données génomiques. Nous pouvons déduire des réseaux d’interactions génétiques à partir de matrices de corrélation ou de co-expression protéine-protéine ou transcription (Shannon et al., 2003 ; Langfelder et Horvath, 2008), et les fusionner avec des analyses de variantes naturelles (Nuzhdin et al., 2012). Nous devrions continuer à développer ces méthodes et d’autres approches afin d’exploiter pleinement nos mesures sur les données génomiques, et de convertir ces mesures quantitatives en analyses de réseau. Si les données génomiques en elles-mêmes ne vont pas fournir la réponse complète à ce qui détermine la capacité d’adaptation de la vie, elles nous permettent de quantifier et d’observer ce qui se passe au niveau moléculaire au cours de l’évolution. Lorsque nous combinons et intégrons cela avec l’hétérogénéité environnementale comme moteur des adaptations, nous pourrons peut-être révéler les mécanismes moléculaires complexes de l’adaptation et de l’évolution.
Déclaration de conflit d’intérêts
L’auteur déclare que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Je remercie Kirsten Jalvingh, Laura Salazar-Jaramillo et Sylvia Gerritsma pour des discussions précieuses. BW a été soutenu par un financement de l’Organisation néerlandaise pour la recherche scientifique (NWO ; subvention 864.08.008).
Aggarwal, K., et Silverman, N. (2008). Régulation positive et négative de la réponse immunitaire de la drosophile. BMB Rep. 41, 267-277. doi : 10.5483/BMBRep.2008.41.4.267
PubMed Abstract | CrossRef Full Text | Google Scholar
Ayroles, J. F., Carbone, M. A., Stone, E. A., Jordan, K. W., Lyman, R. F., Magwire, M. M., et al. (2009). Systems genetics of complex traits in Drosophila melanogaster. Nat. Genet. 41, 299-307. doi : 10.1038/ng.332
PubMed Abstract | CrossRef Full Text | Google Scholar
Brennan, C. A., et Anderson, K. V. (2004). Drosophila : la génétique de la reconnaissance et de la réponse immunitaire innée. Annu. Rev. Immunol. 22, 457-483. doi : 10.1146/annurev.immunol.22.012703.104626
PubMed Abstract | CrossRef Full Text | Google Scholar
Buchmann, K. (2014). Évolution de l’immunité innée : indices des invertébrés aux mammifères en passant par les poissons. Front. Immunol. 5:459. doi : 10.3389/fimmu.2014.00459
PubMed Abstract | CrossRef Full Text | Google Scholar
Carpenter, S., Ricci, E. P., Mercier, B. C., Moore, M. J., et Fitzgerald, K. A. (2014). Régulation post-transcriptionnelle de l’expression des gènes dans l’immunité innée. Nat. Rev. Immunol. 14, 361-376. doi : 10.1038/nri3682
PubMed Abstract | CrossRef Full Text | Google Scholar
Cooper, M. D., et Alder, M. N. (2006). L’évolution des systèmes immunitaires adaptatifs. Cell 124, 815-822. doi : 10.1016/j.cell.2006.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Dobzhansky, T. (1964). La biologie, moléculaire et organismique. Am. Zool. 4, 443-452.
PubMed Abstract | Google Scholar
Drosophila 12 Genomes Consortium, Clark, A. G., Eisen, M. B., Smith, D. R., Bergman, C. M., Oliver, B., et al. (2007). Evolution des gènes et des génomes sur la phylogénie de la drosophile. Nature 450, 203-218. doi : 10.1038/nature06341
PubMed Abstract | CrossRef Full Text | Google Scholar
Edwards, A. C., Ayroles, J. F., Stone, E. A., Carbone, M. A., Lyman, R. F., and Mackay, T. F. C. (2009). Un réseau transcriptionnel associé à la variation naturelle du comportement agressif de la drosophile. Genome Biol. 10, R76. doi : 10.1186/gb-2009-10-7-r76
PubMed Abstract | CrossRef Full Text | Google Scholar
Eslin, P., et Doury, G. (2006). La mouche Drosophila subobscura : un cas naturel de déficit de l’immunité innée. Dev. Comp. Immunol. 30, 977-983. doi : 10.1016/j.dci.2006.02.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Evans, C. J., Hartenstein, V., et Banerjee, U. (2003). Plus épais que le sang : mécanismes conservés dans l’hématopoïèse de la drosophile et des vertébrés. Dev. Cell 5, 673-690. doi : 10.1016/S1534-5807(03)00335-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Fay, J. C., McCullough, H. L., Sniegowski, P. D., and Eisen, M. B. (2004). La variation génétique de la population dans l’expression des gènes est associée à la variation phénotypique dans Saccharomyces cerevisiae. Genome Biol. 5, R26. doi : 10.1186/gb-2004-5-4-r26
PubMed Abstract | CrossRef Full Text | Google Scholar
Fleury, F., Gibert, P., Ris, N., et Allemand, R. (2009). Écologie et évolution du cycle de vie des parasitoïdes frugivores de la drosophile. Adv. Parasitol. 70, 3-44. doi : 10.1016/S0065-308X(09)70001-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Gasch, A. P., Spellman, P. T., Kao, C. M., Carmel-Harel, O., Eisen, M. B., Storz, G., et al. (2000). Programmes d’expression génomique dans la réponse des cellules de levure aux changements environnementaux. Mol. Biol. Cell 11, 4241-4257. doi : 10.1091/mbc.11.12.4241
PubMed Abstract | CrossRef Full Text | Google Scholar
Gerritsma, S., de Haan, A., van de Zande, L., et Wertheim, B. (2013). La variation naturelle des hémocytes différenciés est liée à la résistance aux parasitoïdes chez Drosophila melanogaster. J. Insect Physiol. 59, 148-158. doi : 10.1016/j.jinsphys.2012.09.017
PubMed Abstract | CrossRef Full Text | Google Scholar
Havard, S., Doury, G., Ravallec, M., Brehélin, M., Prevost, G., et Eslin, P. (2012). Caractérisation structurale et fonctionnelle du pseudopodocyte, une cellule immunitaire hirsute produite par deux espèces de drosophiles du groupe obscura. Dev. Comp. Immunol. 36, 323-331. doi : 10.1016/j.dci.2011.05.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Hunt, P., Martinelli, A., Modrzynska, K., Borges, S., Creasey, A., Rodrigues, L., et al. (2010). L’évolution expérimentale, l’analyse génétique et le reséquençage du génome révèlent la mutation conférant la résistance à l’artémisinine dans une lignée isogénique de parasites du paludisme. BMC Genomics 11:499. doi : 10.1186/1471-2164-11-499
PubMed Abstract | CrossRef Full Text | Google Scholar
Ivanov, P., et Anderson, P. (2013). Réseaux de régulation post-transcriptionnelle dans l’immunité. Immunol. Rev. 253, 253-272. doi : 10.1111/imr.12051
PubMed Abstract | CrossRef Full Text | Google Scholar
Jalvingh, K. M., Chang, P. L., Nuzhdin, S. V., et Wertheim, B. (2014). Changements génomiques sous évolution rapide : sélection pour la résistance aux parasitoïdes. Proc. Biol. Sci. 281, 20132303. doi : 10.1098/rspb.2013.2303
PubMed Abstract | CrossRef Full Text | Google Scholar
Jones, F. C., Grabherr, M. G., Chan, Y. F., Russell, P., Mauceli, E., Johnson, J., et al. (2012). The genomic basis of adaptive evolution in threespine sticklebacks. Nature 484, 55-61. doi : 10.1038/nature10944
PubMed Abstract | CrossRef Full Text | Google Scholar
Knight, C. G., Zitzmann, N., Prabhakar, S., Antrobus, R., Dwek, R., Hebestreit, H., et al. (2006). Unraveling adaptive evolution : how a single point mutation affects the protein coregulation network. Nat. Genet. 38, 1015. doi : 10.1038/ng1867
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., et Godfray, H. C. J. (1999). Modèles géographiques dans l’évolution de la résistance et de la virulence chez la drosophile et ses parasitoïdes. Am. Nat. 153, S61-S74. doi : 10.1086/303212
CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., Ferrari, J., et Godfray, H. C. J. (2002). Coûts de la résistance dans les interactions insecte-parasite et insecte-parasitoïde. Parasitology 125(Suppl.), S71-S82. doi : 10.1017/S0031182002001750
PubMed Abstract | CrossRef Full Text | Google Scholar
Kraaijeveld, A. R., et van Alphen, J. J. M. (1995). Variation géographique de la capacité d’encapsulation des larves de Drosophila melanogaster et preuve de composants spécifiques aux parasitoïdes. Evol. Ecol. 9, 10-17. doi : 10.1007/BF01237692
CrossRef Full Text | Google Scholar
Langfelder, P., et Horvath, S. (2008). WGCNA : un paquet R pour l’analyse de réseau de corrélation pondérée. BMC Bioinformatics 9:559. doi : 10.1186/1471-2105-9-559
PubMed Abstract | CrossRef Full Text | Google Scholar
Lavine, M. D., et Strand, M. R. (2002). Les hémocytes des insectes et leur rôle dans l’immunité. Insect Biochem. Mol. Biol. 32, 1295-1309. doi : 10.1016/S0965-1748(02)00092-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., and Little, T. J. (2009). L’immunité dans un monde variable. Philos. Trans. R. Soc. Lond. B Biol. Sci. 364, 15-26. doi : 10.1098/rstb.2008.0141
PubMed Abstract | CrossRef Full Text | Google Scholar
Lazzaro, B. P., et Schneider, D. S. (2014). La génétique de l’immunité. G3 (Bethesda) 4, 943-945. doi : 10.1534/g3.114.011684
PubMed Abstract | CrossRef Full Text | Google Scholar
Lehner, B. (2013). Du génotype au phénotype : leçons des organismes modèles pour la génétique humaine. Nat. Rev. Genet. 14, 168-178. doi : 10.1038/nrg3404
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemaitre, B., et Hoffmann, J. (2007). La défense de l’hôte de Drosophila melanogaster. Annu. Rev. Immunol. 25, 697-743. doi : 10.1146/annurev.immunol.25.022106.141615
PubMed Abstract | CrossRef Full Text | Google Scholar
Liew, F. Y., Xu, D., Brint, E. K., et O’Neill, L. A. J. (2005). Régulation négative des réponses immunitaires médiées par les récepteurs Toll-like. Nat. Rev. Immunol. 5, 446-458. doi : 10.1038/nri1630
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnen, C. R., Poh, Y.-P., Peterson, B. K., Barrett, R. D. H., Larson, J. G., Jensen, J. D., et al. (2013). L’évolution adaptative de plusieurs traits par des mutations multiples sur un seul gène. Science 339, 1312-1316. doi : 10.1126/science.1233213
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackay, T. F., et Moore, J. H. (2014). Pourquoi l’épistasie est importante pour aborder la génétique complexe des maladies humaines. Genome Med. 6, 42. doi : 10.1186/gm561
PubMed Abstract | CrossRef Full Text | Google Scholar
Manolio, T. A., Collins, F. S., Cox, N. J., Goldstein, D. B., Hindorff, L. A., Hunter, D. J., et al. (2009). Finding the missing heritability of complex diseases (Trouver l’héritabilité manquante des maladies complexes). Nature 461, 747-753. doi : 10.1038/nature08494
PubMed Abstract | CrossRef Full Text | Google Scholar
Márkus, R., Lerner, Z., Honti, V., Csordás, G., Zsámboki, J., Cinege, G., et al. (2015). Les hémocytes géants multinucléés sont des cellules effectrices dans les réponses immunitaires à médiation cellulaire de la drosophile. J. Innate Immun. doi : 10.1159/000369618 .
PubMed Abstract | CrossRef Full Text | Google Scholar
Moore, J. H., et Williams, S. M. (eds). (2015). Epistasis. New York, NY : Springer. doi : 10.1007/978-1-4939-2155-3
CrossRef Full Text | Google Scholar
Nagy, A., Perrimon, N., Sandmeyer, S., et Plasterk, R. (2003). Tailoring the genome : the power of genetic approaches. Nat. Genet. 33(Suppl.), 276-284. doi : 10.1038/ng1115
PubMed Abstract | CrossRef Full Text | Google Scholar
Nowick, K., Gernat, T., Almaas, E., et Stubbs, L. (2009). Les différences dans les modèles d’expression des gènes humains et chimpanzés définissent un réseau évolutif de facteurs de transcription dans le cerveau. Proc. Natl. Acad. Sci. U.S.A. 106, 22358-22363. doi : 10.1073/pnas.0911376106
PubMed Abstract | CrossRef Full Text | Google Scholar
Nuzhdin, S. V., Friesen, M. L., et McIntyre, L. M. (2012). Cartographie génotype-phénotype dans un monde post-GWAS. Trends Genet. 28, 421-426. doi : 10.1016/j.tig.2012.06.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Obbard, D. J., Welch, J. J., Kim, K.-W., et Jiggins, F. M. (2009). Quantifier l’évolution adaptative dans le système immunitaire de la drosophile. PLoS Genet. 5:e1000698. doi : 10.1371/journal.pgen.1000698
PubMed Abstract | CrossRef Full Text | Google Scholar
Papp, B., Notebaart, R. A., et Pál, C. (2011). Approches de biologie systémique pour la prédiction de l’évolution génomique. Nat. Rev. Genet. 12, 591-602. doi : 10.1038/nrg3033
PubMed Abstract | CrossRef Full Text | Google Scholar
Pedra, J. H. F., McIntyre, L. M., Scharf, M. E., and Pittendrigh, B. R. (2004). Profil de transcription à l’échelle du génome de la drosophile résistante au dichlorodiphényltrichloroéthane (DDT) sélectionnée sur le terrain et en laboratoire. Proc. Natl. Acad. Sci. U.S.A. 101, 7034-7039. doi : 10.1073/pnas.0400580101
PubMed Abstract | CrossRef Full Text | Google Scholar
Phillips, P. C. (2008). Epistasis-le rôle essentiel des interactions entre les gènes dans la structure et l’évolution des systèmes génétiques. Nat. Rev. Genet. 9, 855-867. doi : 10.1038/nrg2452
PubMed Abstract | CrossRef Full Text | Google Scholar
Rolff, J., et Siva-Jothy, M. T. (2003). L’immunologie écologique des invertébrés. Science 301, 472-475. doi : 10.1126/science.1080623
PubMed Abstract | CrossRef Full Text | Google Scholar
Sackton, T. B., Lazzaro, B. P., Schlenke, T. A., Evans, J. D., Hultmark, D., and Clark, A. G. (2007). Dynamic evolution of the innate immune system in Drosophila. Nat. Genet. 39, 1461-1468. doi : 10.1038/ng.2007.60
PubMed Abstract | CrossRef Full Text | Google Scholar
Salazar-Jaramillo, L., Paspati, A., van de Zande, L., Vermeulen, C. J., Schwander, T., et Wertheim, B. (2014). Évolution d’une réponse immunitaire cellulaire chez la drosophile : une analyse comparative phénotypique et génomique. Genome Biol. Evol. 6, 273-289. doi : 10.1093/gbe/evu012
PubMed Abstract | CrossRef Full Text | Google Scholar
Schadt, E. E. (2009). Les réseaux moléculaires comme capteurs et conducteurs de maladies humaines communes. Nature 461, 218-223. doi : 10.1038/nature08454
PubMed Abstract | CrossRef Full Text | Google Scholar
Schlenke, T. A., Morales, J., Govind, S., and Clark, A. G. (2007). Contrastant les stratégies d’infection dans les parasitoïdes de guêpe généralistes et spécialistes de Drosophila melanogaster. PLoS Pathog. 3:e158. doi : 10.1371/journal.ppat.0030158
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2003). La variation de la défense immunitaire comme une question d’écologie évolutive. Proc. R. Soc. B Biol. Sci. 270, 357-366. doi : 10.1098/rspb.2002.2265
PubMed Abstract | CrossRef Full Text | Google Scholar
Schmid-Hempel, P. (2005). Écologie évolutive des défenses immunitaires des insectes. Annu. Rev. Entomol. 50, 529-551. doi : 10.1146/annurev.ento.50.071803.130420
PubMed Abstract | CrossRef Full Text | Google Scholar
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al. (2003). Cytoscape : un environnement logiciel pour les modèles intégrés de réseaux d’interactions biomoléculaires. Genome Res. 13, 2498-2504. doi : 10.1101/gr.1239303
PubMed Abstract | CrossRef Full Text | Google Scholar
Silverman, N., et Maniatis, T. (2001). Les voies de signalisation NF-κB dans l’immunité innée des mammifères et des insectes. Genes Dev. 15, 2321-2342. doi : 10.1101/gad.909001
PubMed Abstract | CrossRef Full Text | Google Scholar
Sironi, M., Cagliani, R., Forni, D., et Clerici, M. (2015). Aperçus évolutifs sur les interactions hôte-pathogène à partir de données de séquences de mammifères. Nat. Rev. Genet. 16, 224-236. doi : 10.1038/nrg3905
PubMed Abstract | CrossRef Full Text | Google Scholar
Stapley, J., Reger, J., Feulner, P. G. D., Smadja, C., Galindo, J., Ekblom, R., et al. (2010). Adaptation genomics : the next generation. Trends Ecol. Evol. 25, 705-712. doi : 10.1016/j.tree.2010.09.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Star, B., Nederbragt, A. J., Jentoft, S., Grimholt, U., Malmstrøm, M., Gregers, T. F., et al. (2011). La séquence du génome de la morue de l’Atlantique révèle un système immunitaire unique. Nature 477, 207-210. doi : 10.1038/nature10342
PubMed Abstract | CrossRef Full Text | Google Scholar
Stearns, F. W. (2010). Cent ans de pléiotropie : une rétrospective. Genetics 186, 767-773. doi : 10.1534/genetics.110.122549
PubMed Abstract | CrossRef Full Text | Google Scholar
Storz, J. F., et Wheat, C. W. (2010). Intégrer les approches évolutives et fonctionnelles pour inférer l’adaptation à des loci spécifiques. Evolution 64, 2489-2509. doi : 10.1111/j.1558-5646.2010.01044.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Tenaillon, O., Rodríguez-Verdugo, A., Gaut, R. L., McDonald, P., Bennett, A. F., Long, A. D., et al. (2012). La diversité moléculaire de la convergence adaptative. Science 335, 457-461. doi : 10.1126/science.1212986
PubMed Abstract | CrossRef Full Text | Google Scholar
Turner, T. L., Stewart, A. D., Fields, A. T., Rice, W. R., and Tarone, A. M. (2011). Le reséquençage des populations évoluées expérimentalement révèle la base génétique de la variation de la taille du corps chez Drosophila melanogaster. PLoS Genet. 7:e1001336. doi : 10.1371/journal.pgen.1001336
PubMed Abstract | CrossRef Full Text | Google Scholar
Wagner, G. P., et Altenberg, L. (1996). Les adaptations complexes et l’évolution de l’évolutivité. Evolution 50, 967-976. doi : 10.2307/2410639
CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Hopkins, M. G., Walther Boer, M., et Godfray, H. C. (2011). Génomique fonctionnelle de l’évolution de la résistance accrue au parasitisme chez la drosophile. Mol. Ecol. 20, 932-949. doi : 10.1111/j.1365-294X.2010.04911.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Wertheim, B., Kraaijeveld, A. R., Schuster, E., Blanc, E., Hopkins, M., Pletcher, S. D., et al. (2005). Genome-wide gene expression in response to parasitoid attack in Drosophila. Genome Biol. 11, R94. doi : 10.1186/gb-2005-6-11-r94
PubMed Abstract | CrossRef Full Text | Google Scholar
Zak, D. E., Tam, V. C., et Aderem, A. (2014). Analyse de l’immunité innée au niveau des systèmes. Annu. Rev. Immunol. 32, 547-577. doi : 10.1146/annurev-immunol-032713-120254
PubMed Abstract | CrossRef Full Text | Google Scholar