Wie verhindert die DNA-Polymerase Mutationen
Mutationen sind dauerhafte Veränderungen der Nukleotidsequenz eines bestimmten Organismus. Sie können durch Fehler bei der DNA-Replikation oder durch externe Mutagene entstehen. Die Wirkung einer Mutation kann für die Zelle entweder vorteilhaft oder nachteilig sein. Die Zellen verfügen jedoch über verschiedene Mechanismen, um Mutationen zu verhindern. Die DNA-Polymerase, das an der DNA-Replikation beteiligte Enzym, ist mit mehreren Mechanismen ausgestattet, um Fehler während der DNA-Replikation zu verhindern. Während der DNA-Replikation werden die fehlgepaarten Basen durch Korrekturlesen ersetzt. Unmittelbar nach der DNA-Replikation werden die verbleibenden fehlgepaarten Basen durch stranggerichtete Mismatch-Reparatur ersetzt. Darüber hinaus werden die durch externe Faktoren verursachten Mutationen durch verschiedene Mechanismen wie Exzisionsreparatur, chemische Umkehrung und Doppelstrangbruchreparatur repariert. Wenn der Schaden reversibel ist, wird die Zelle der Apoptose unterworfen, um zu verhindern, dass die fehlerhafte DNA an die Nachkommen weitergegeben wird.
Behandelte Schlüsselbereiche
1. Was ist eine Mutation
– Definition, Arten, Ursachen
2. Wie verhindert die DNA-Polymerase Mutationen
– Korrekturlesen, Strang-gerichtete Fehlpaarungsreparatur
Schlüsselbegriffe: DNA-Polymerase, Strand-Directed Mismatch Repair, Mut-Proteine, Mutation, Proofreading

Was ist eine Mutation
Eine Mutation ist eine dauerhafte und vererbbare Veränderung in der Nukleotidsequenz des Genoms. Mutationen können durch Fehler bei der DNA-Replikation oder durch äußere Faktoren, so genannte Mutagene, entstehen. Die drei Formen von Mutationen sind Punktmutationen, Frameshift-Mutationen und chromosomale Mutationen.
Punktmutationen
Punktmutationen sind einzelne Nukleotidsubstitutionen. Die drei Arten von Punktmutationen sind Missense-, Nonsense- und Silent-Mutationen. Bei der Missense-Mutation wird ein einzelnes Codon des Gens verändert, wodurch die Aminosäure in der Polypeptidkette verändert wird. Nonsense-Mutationen verändern zwar die Codon-Sequenz, nicht aber die Aminosäuresequenz. Stille Mutationen verändern ein einzelnes Codon zu einem anderen Codon, das für dieselbe Aminosäure steht. Punktmutationen werden durch Fehler bei der DNA-Replikation und durch Mutagene verursacht. Verschiedene Arten von Punktmutationen sind in Abbildung 1 dargestellt.
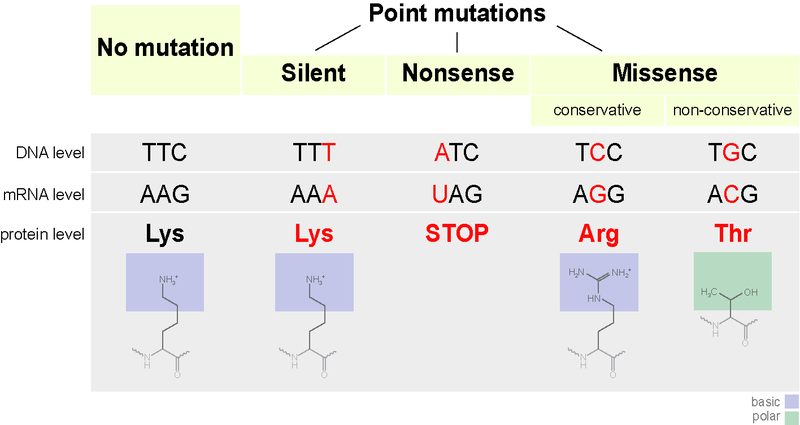
Abbildung 1: Punktmutationen
Rahmenverschiebungsmutationen
Rahmenverschiebungsmutationen sind Einfügungen oder Streichungen einzelner oder mehrerer Nukleotide im Genom. Insertionen, Deletionen und Duplikationen sind die drei Arten von Frameshift-Mutationen. Insertionen sind das Hinzufügen von einem oder mehreren Nukleotiden zur Sequenz, während Deletionen das Entfernen von mehreren Nukleotiden aus der Sequenz bedeuten. Duplikationen sind die Wiederholung von mehreren Nukleotiden. Frameshift-Mutationen werden auch durch Fehler bei der DNA-Replikation und durch Mutagene verursacht.
Chromosomale Mutationen
Chromosomale Mutationen sind Veränderungen von Chromosomenabschnitten. Zu den Arten von Chromosomenmutationen gehören Translokationen, Genduplikationen, intrachromosomale Deletionen, Inversionen und der Verlust der Heterozygotie. Translokationen sind der Austausch von Chromosomenabschnitten zwischen nicht-homologen Chromosomen. Bei der Genduplikation können mehrere Kopien eines bestimmten Allels auftreten, wodurch die Gendosis erhöht wird. Die Entfernung von Chromosomenabschnitten wird als intrachromosomale Deletion bezeichnet. Bei Inversionen ändert sich die Ausrichtung eines Chromosomenabschnitts. Die Heterozygotie eines Gens kann durch den Verlust eines Allels auf einem Chromosom durch Deletion oder genetische Rekombination verloren gehen. Chromosomenmutationen werden hauptsächlich durch externe Mutagene und durch mechanische Schäden an der DNA verursacht.
Wie verhindert die DNA-Polymerase Mutationen
Die DNA-Polymerase ist das Enzym, das während der DNA-Replikation für das Anfügen von Nukleotidbasen an den wachsenden Strang verantwortlich ist. Da die Nukleotidsequenz eines Genoms die Entwicklung und Funktionsweise eines bestimmten Organismus bestimmt, ist es unerlässlich, während der DNA-Replikation eine exakte Kopie des vorhandenen Genoms zu synthetisieren. Im Allgemeinen behält die DNA-Polymerase während der DNA-Replikation eine hohe Zuverlässigkeit bei und baut nur ein einziges falsches Nukleotid pro 109 hinzugefügte Nukleotide ein. Tritt also eine Fehlpaarung zwischen stickstoffhaltigen Basen zusätzlich zu den standardmäßigen komplementären Basenpaaren auf, fügt die DNA-Polymerase dieses Nukleotid an die wachsende Kette an, wodurch eine häufige Mutation entsteht. Die Fehler der DNA-Replikation werden durch zwei Mechanismen korrigiert, die als Korrekturlesen und stranggerichtete Fehlpaarungsreparatur bekannt sind.
Korrekturlesen
Korrekturlesen bezieht sich auf einen ersten Mechanismus zur Korrektur der fehlenden Basenpaare des wachsenden DNA-Strangs und wird von der DNA-Polymerase durchgeführt. Die DNA-Polymerase führt die Korrekturlesung in zwei Schritten durch. Die erste Korrektur erfolgt unmittelbar vor dem Anfügen eines neuen Nukleotids an die wachsende Kette. Die Affinität der richtigen Nukleotide für die DNA-Polymerase ist um ein Vielfaches höher als die der falschen Nukleotide. Das Enzym sollte jedoch kurz nach der Bindung des ankommenden Nukleotids an die Matrize durch Wasserstoffbrückenbindungen, aber vor der Bindung des Nukleotids an den wachsenden Strang durch die DNA-Polymerase eine Konformationsänderung erfahren. Die falsch basengepaarten Nukleotide neigen dazu, während der Konformationsänderung der DNA-Polymerase von der Matrize zu dissoziieren. Daher ermöglicht dieser Schritt der DNA-Polymerase, das Nukleotid doppelt zu überprüfen, bevor es dauerhaft an den wachsenden Strang angehängt wird. Der Korrekturlesemechanismus der DNA-Polymerase ist in Abbildung 2 dargestellt.
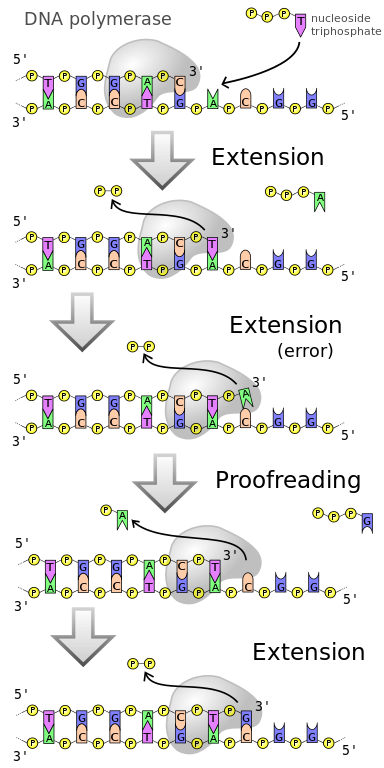
Abbildung 2: Korrekturlesen
Der zweite Korrekturleseschritt ist als exonukleolytisches Korrekturlesen bekannt. Er erfolgt unmittelbar nach dem Einbau eines fehlangepassten Nukleotids in den wachsenden Strang, was selten vorkommt. Die DNA-Polymerase ist nicht in der Lage, das zweite Nukleotid neben dem fehlangepassten Nukleotid einzufügen. Eine separate katalytische Stelle der DNA-Polymerase, die als 3′- bis 5′-Proofreading-Exonuklease bekannt ist, verdaut die fehlgepaarten Nukleotide aus der wachsenden Kette.
Strand-Directed Mismatch Repair
Trotz Proofreading-Mechanismen kann die DNA-Polymerase während der DNA-Replikation immer noch falsche Nukleotide in den wachsenden Strang einbauen. Die Replikationsfehler, die dem Korrekturlesen entgangen sind, werden durch die Strang-gerichtete Fehlpaarungsreparatur beseitigt. Dieses System erkennt ein Verzerrungspotenzial in der DNA-Helix, das auf nicht übereinstimmende Basenpaare zurückzuführen ist. Das Reparatursystem sollte jedoch die falsche Base von der vorhandenen Base unterscheiden, bevor es die Fehlpaarung ersetzt. Im Allgemeinen ist E. coli auf das DNA-Methylierungssystem angewiesen, um den alten DNA-Strang in der Doppelhelix zu erkennen, da der neu synthetisierte Strang möglicherweise nicht so schnell einer DNA-Methylierung unterzogen wird. In E. coli ist der A-Rest des GATC methyliert. Die Treue der DNA-Replikation wird durch die Wirkung des stranggerichteten Mismatch-Reparatursystems um einen zusätzlichen Faktor 102 erhöht. Die Wege der DNA-Fehlpaarungsreparatur bei Eukaryoten, Bakterien und E. coli sind in Abbildung 3 dargestellt.
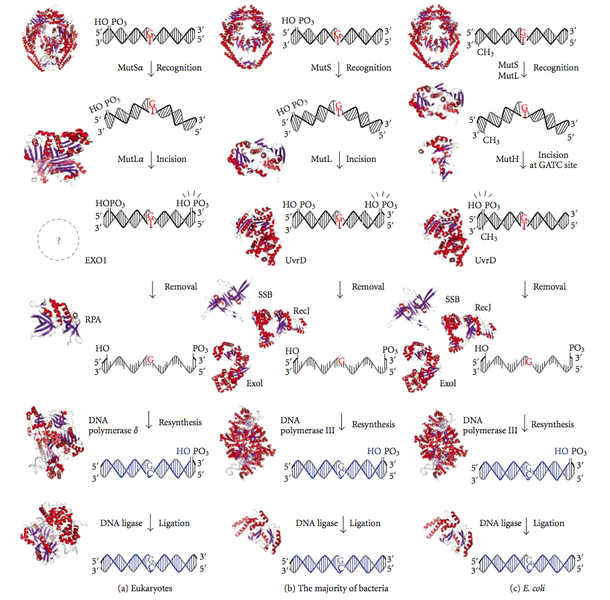
Abbildung 3: DNA-Fehlpaarungsreparatur bei Eukaryoten, Bakterien und E. coli
Bei der stranggerichteten Fehlpaarungsreparatur bewegen sich drei komplexe Proteine durch den neu synthetisierten DNA-Strang. Das erste Protein mit der Bezeichnung MutS erkennt die Verzerrungen in der DNA-Doppelhelix und bindet daran. Das zweite Protein, MutL, erkennt und bindet an MutS, wodurch das dritte Protein, MutH, angelockt wird, das den unmethylierten oder den neu synthetisierten Strang unterscheidet. Nach der Bindung schneidet das MutH den unmethylierten DNA-Strang unmittelbar vor dem G-Rest in der GATC-Sequenz. Eine Exonuklease ist für den Abbau des Strangs stromabwärts der Fehlpaarung verantwortlich. Dieses System degradiert jedoch Regionen mit weniger als 10 Nukleotiden, die von der DNA-Polymerase 1 leicht wieder synthetisiert werden können. Die Mut-Proteine der Eukaryonten sind homolog zu denen von E. coli.
Schlussfolgerung
Mutationen sind dauerhafte Veränderungen der Nukleotidsequenz des Genoms, die durch Fehler bei der DNA-Replikation oder durch die Wirkung externer Mutagene entstehen können. Die Fehler bei der DNA-Replikation können durch zwei Mechanismen korrigiert werden, die als Korrekturlesen und stranggerichtete Mismatch-Reparatur bekannt sind. Die Korrekturlesung wird von der DNA-Polymerase selbst während der DNA-Synthese durchgeführt. Die stranggerichtete Fehlpaarungsreparatur wird von Mut-Proteinen unmittelbar nach der DNA-Replikation durchgeführt. Diese Reparaturmechanismen sind jedoch an der Erhaltung der Integrität des Genoms beteiligt.
Referenz:
1. Alberts, Bruce. „DNA Replication Mechanisms.“ Molecular Biology of the Cell. 4. Auflage, U.S. National Library of Medicine, 1. Januar 1970, hier verfügbar.
2. Brown, Terence A. „Mutation, Reparatur und Rekombination“. Genomes. 2nd edition, U.S. National Library of Medicine, 1 Jan. 1970, Available here.
Image Courtesy:
1. „Different Types of Mutations“ By Jonsta247 – This file was derived from:Punktmutationen-de.png (GFDL) via Commons Wikimedia
2. „DNA polymerase“ By I, Madprime (CC BY-SA 3.0) via Commons Wikimedia
3. „DNA mismatch repair“ By Kenji Fukui – (CC BY 3.0) via Commons Wikimedia
![]()